【文献速递】通过宏基因组组装基因组揭示两种不同酱油发酵过程中产生生物胺的微生物的见解
- 看不见的线
- 564
- 2025-11-06 10:01:12
- 原创
文章题目:Insights into the biogenic amine-generating microbes during two different types of soy sauce fermentation as revealed by metagenome-assembled genomes
文章链接:https://doi.org/10.26599/FSHW.2024.9250064
发表时间:2025-03-18
期刊:Food Science and Human Wellness
影响因子:7.46
一、研究背景与核心问题
生物胺(BAs)是发酵食品中由微生物通过氨基酸脱羧产生的有害化合物,过量摄入可能导致头痛、高血压等健康风险。酱油作为传统发酵食品,其JP(日式)和CP(广式)工艺在原料配比、发酵条件(温度、盐度、接种酵母)上存在显著差异,但两者发酵过程中BAs生成微生物的多样性及功能尚不明确。
这项研究基于宏基因组数据比较JP与CP工艺下BAs的分布差异及关键产胺微生物;解析微生物群落中BAs合成基因的丰度特征;通过宏基因组组装基因组(MAGs)鉴定产胺微生物的物种和功能。
- 二、材料与方法
样品采集:从工业酱油厂获取JP和CP工艺发酵7、15、30、60、90、120天的酱醪样品(各阶段生物重复混合)。
理化分析:测定pH、盐度(NaCl)、总酸度(TA)、氨基态氮(AAN)及8种BAs含量(腐胺、酪胺、组胺等)。
宏基因组测序:
·DNA提取、文库构建(Illumina NovaSeq 6000平台,2×150 bp);
·序列组装(IDBA-UD)、基因预测(MetaGeneMark)、去冗余(CD-HIT);
·物种注释(DIAMOND比对NCBI-NR库);
·BAs合成基因注释(KEGG数据库,阈值:氨基酸相似性>30%,覆盖度>95%)。
MAGs重建与分析:
·基因组分箱(CONCOCT)、质量评估(CheckM,完整性>80%,污染<10%);
·物种分类(GTDB-Tk)、功能注释(Prodigal、KofamScan);
·毒力基因鉴定(VFDB数据库,针对肺炎克雷伯菌)。
统计分析:
·差异显著性(ANOVA、t检验)、相关性分析(Spearman);
·微生物-BAs基因共现网络(Cytoscape,|ρ|≥0.6,P<0.05)。
- 三、研究结果
- 醪醋发酵过程中的理化特性和BA概况
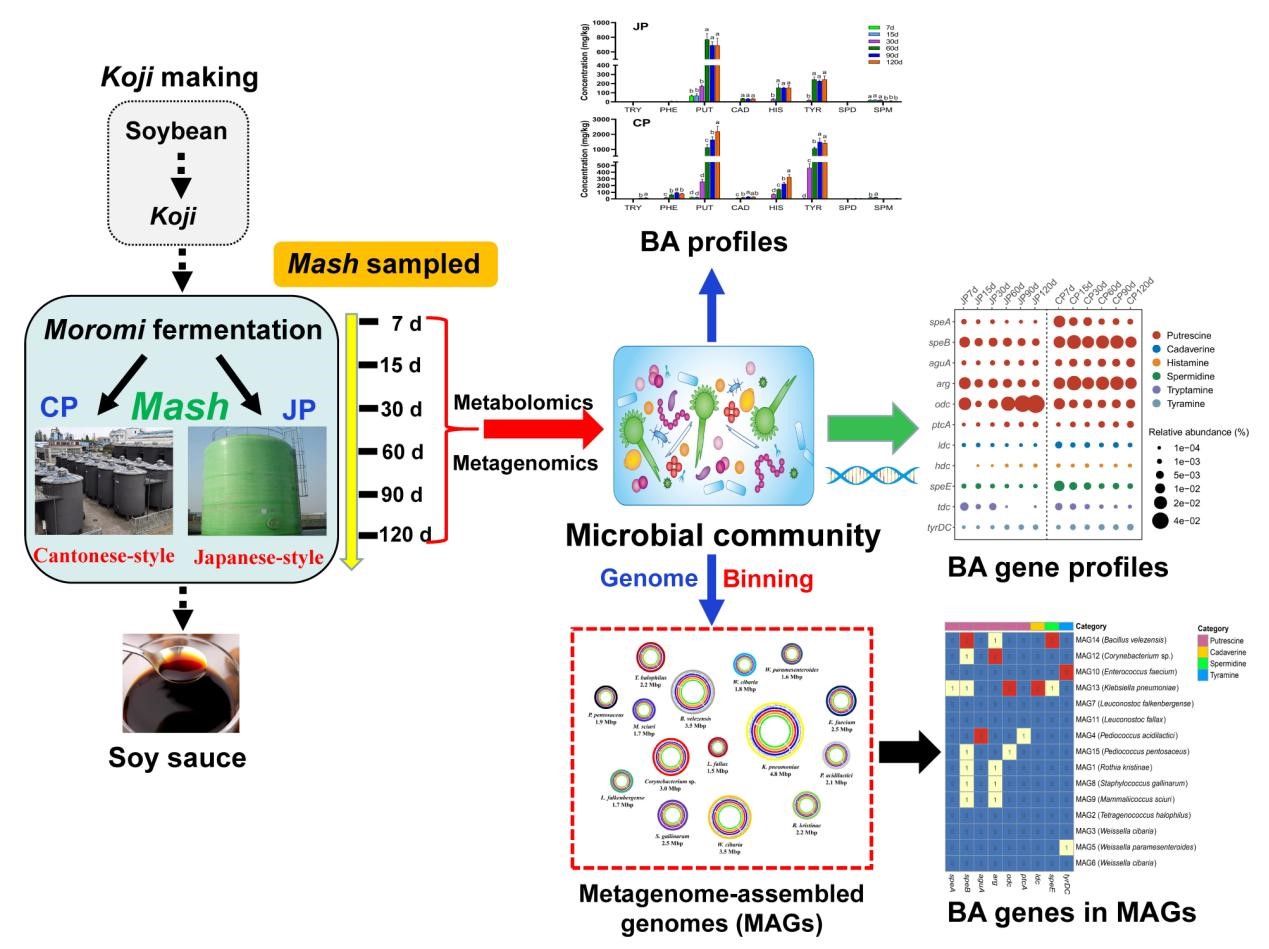
BA分析表明,腐胺、组胺和酪胺是两种发酵过程中的主要BAs,但不同BAs含量的变化趋势不同(图1)。在JP发酵过程中,腐胺在后期三个阶段是最丰富的BA物种(717.22 mg/kg),其次是酪胺(239.46 mg/kg)和组胺(154.69 mg/kg)(图1A)。然而,在初始阶段(发酵7~15 d)含量急剧增加后,这三个BA的含量在后期三个阶段保持稳定。此外,还检测到尸胺,发酵结束时含量为(34.54±1.83)mg/kg。但精氨酸含量从21.37 mg/kg下降到8.19 mg/kg(P<0.05)。发酵过程中未检测到色氨酸。CP发酵过程中,三种BAs(腐胺、酪胺和组胺)的浓度从发酵初期迅速增加(P<0.05)(图1B)。发酵结束时(120 d)腐胺、酪胺和组胺的含量分别为(2200.48±350.73)、(1432.07±151.20)和(327.51±40.11)mg/kg。与JP发酵相比,CP发酵过程中苯乙胺的含量较高。酪胺的含量与先前的研究相当,在醪醋发酵的第三个月,酪胺浓度增加到2073.27 mg/kg。一些研究建议对食品中的BA含量进行限制。建议食品中总钡含量为1000 mg/kg,组胺含量为100 mg/kg,酪胺含量为100~800 mg/kg,β-苯乙胺含量为30 mg/kg。此外,本研究还比较了发酵结束时(120 d)8个BA之间的差异。在检测到的8种BAs中,CP中的5种BAs(腐胺、酪胺、组胺、苯乙胺和色氨酸)的含量明显高于JP中的含量(P<0.05)(图1C)。两种不同发酵工艺中BA浓度的明显差异可归因于环境因素,特别是温度、氧气可用性、光周期和盐度,以及起始物培养物(含或不含起始物鲁氏接合酵母),这些因素在JP和CP制造工艺中有所不同。
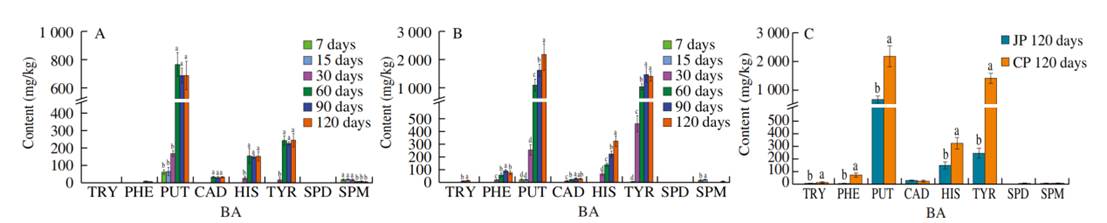
图1 在发酵过程中从(A)JP酱油和(B)CP酱油的酱油醪液样本中鉴定的BA谱图;(C)在两种不同发酵过程中120 d发酵后醪液样本中BA的比较
- 不同酱油发酵类型的微生物群落特征变化
从属的角度来看,四联球菌属、葡萄球菌和魏斯氏菌与真菌曲霉一起主导了JP和CP群落(图2),这与之前在酱油样本中的一项研究结果相似。在这些属中,随着发酵的进行,四联球菌属的丰度急剧增加(在JP的所有序列中从0.02%增加到59.2%,在CP中从0.01%增加到36.60%),但在JP样本中,四联球菌属的相对丰度较高(发酵60~120 d)。作为嗜盐性实验室,四联球菌属已被确定为酱油中的主要微生物。相比之下,在所有发酵阶段,CP中葡萄球菌的丰度都高于JP中,平均丰度分别为32.6%和8.5%(图2)。然而,JP和CP样品中魏斯氏菌在发酵15 d后丰度均呈下降趋势,表明魏斯氏菌不耐盐。同样,在整个发酵过程中,曲霉菌种群数量下降(在JP中从32.1%降至0.12%,在CP中从17.1%降至0.65%)(图2)。发酵前阶段较高的曲霉相对丰度来源于曲霉,其中曲霉菌种被用作起始培养物。此外,在CP群落中,芽孢杆菌的丰度(平均3.16%)高于JP群落(0.22%)。其他真菌的丰度相对较低,如酵母菌共聚糖酵母、毕赤酵母、念珠菌、克鲁维酵母、拟威克酵母属和酿酒酵母。
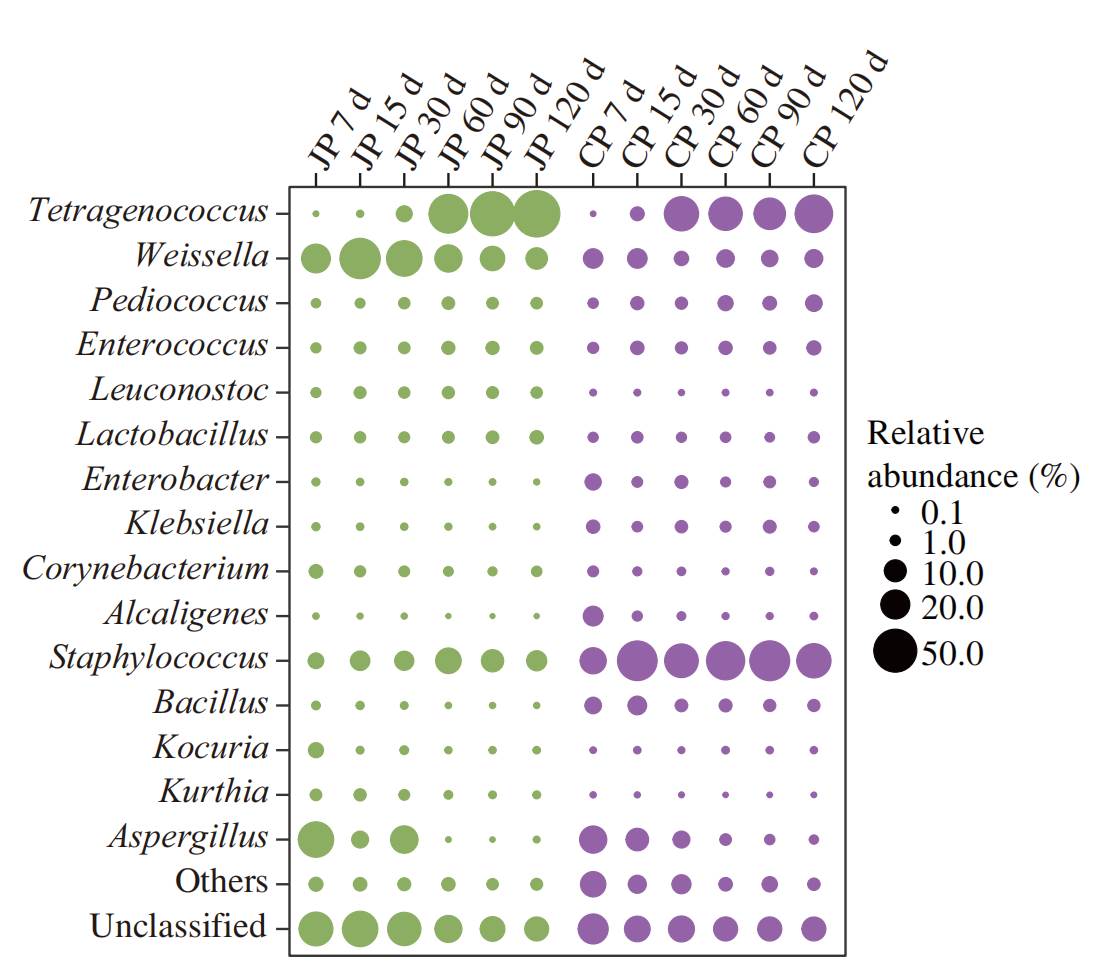
图2 在两个不同的发酵过程中,15个最丰富属的微生物分类组成在属一级的分布情况
- BA基因的概况及其与醪液化学和生物体的关系
特定基因的丰度是根据相对丰度计算的。检测到的参与腐胺合成的基因包括两个脱羧酶基因(精氨酸脱羧酶,speA;和鸟氨酸脱羧酶,odc)和四个非脱羧酶基因(胍基丁胺酶,speB;精氨酸脱亚胺酶,aguA;精氨酸酶,arg;和腐胺氨基甲酰转移酶,ptcA)(图3)。这些基因表现出不同的丰度特征。基因speB、arg和odc是JP中含量最高的基因,而基因speB、arg和odc以及speA在CP中占主导地位。其中,JP中ODC基因的相对丰度在发酵15 d后从2.6×10-5增加到4.9×10-4,在发酵结束时(120 d)丰度最高(4.9×10-4)。在JP和CP发酵过程中,ptcA基因的丰度增加,丰度分别从2.1×10-6增加到1.7×10-5,从5.9×10-6增加到了3.3×10-5。然而,在整个发酵过程中,speA基因的丰度急剧下降(在JP中从1.4×10-5降至2.2×10-6,在CP中从1.6×10-4降至1.8×10-5)。此外,参与酪胺生成的酪氨酸脱羧酶基因(tyrDC)丰度在每个发酵时间点上都增加了(在JP中从1.8×10-6增加到1.4×10-5,在CP中从6.7×10-6增加到了2.8×10-5),并且在CP中比在JP中高(平均为3.3倍)。因此,推测CP混合物样品中检测到的较高浓度的酪胺(图1)与酪胺生成物中酪胺酮C的高表达水平有关。最后,还鉴定了编码产生组胺(hdc)和色氨酸(tdc)的杂环胺脱羧酶的基因(图3)。其中,hdc基因在JP中的丰度相对较低,平均为1.3×10-6,在CP中为3.3×10-6。此外,还检测到一个参与尸胺生成的赖氨酸脱羧酶基因(ldc),其在JP中的平均丰度为3.5×10-6,在CP中的平均丰度的1.7×10-5。CP的speE(编码精胺合酶)基因丰度高于JP。然而,未检测到编码精氨酸合酶和编码苯丙氨酸脱羧酶用于苯乙胺合成的基因。虽然在本研究中未发现pdcA基因,但在60~120 d的发酵周期中,JP中的苯乙胺平均为6.68 mg/kg,CP中的苯乙酰胺平均为78.38 mg/kg。
图3 在两种不同的酱油发酵过程中参与生物胺生成的基因的相对丰度
- 基因组重建和MAG丰度
在连续筛选和基因组质量控制后,共重建了JP和CP样品中混合质粒组中的44个MAG,其中15个基因组的完整性在80%以上,污染少于10%(图4)。这15个基因组的基因组大小在1487942~4833112 bp之间,平均为2427150 bp;每个基因组的连续序列数在60到548之间;每个基因的ORF数在1502到4829之间。
FastANI被用于进行分类学归属并评估MAG的新颖性,结果显示一个MAG与NCBI数据库中已知的原核生物基因组之间的相似度较低(<95%)。新发现的物种(MAG12)被鉴定为棒状杆菌属,属于棒状杆菌科家族,与棒状杆菌科变种聚类,相似度达到85.9%(图4)。需要指出的是,虽然发酵过程中存在较高丰度的某些曲霉属物种,但本研究并未完整检索到真核生物基因组,因为这些物种的基因组通常较大,从宏基因组数据集中重构较为困难。在JP样品的发酵初期阶段(7~30 d),W. cibaria的丰度显著较高,但在中期至后期阶段(60~120 d)其丰度略有下降。
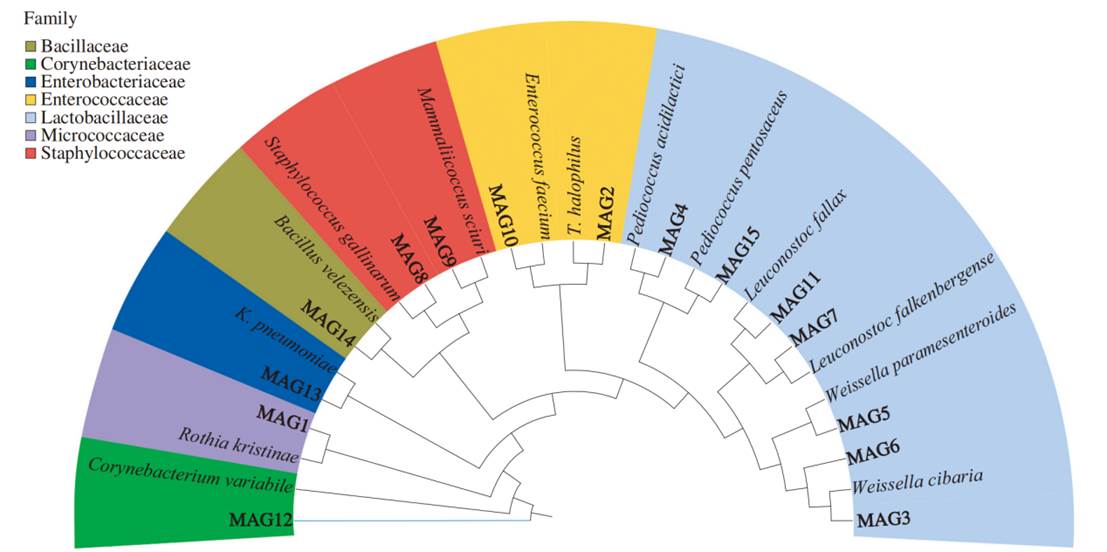
图4 从酱油泥状代谢组学数据中重构的精选MAGs的属分类归属
- BA基因在MAG中的分布模式
本研究的元基因组组装、基因组筛选和注释使我们能够区分酱油发酵微生物组中携带各种BA基因的宿主。研究发现,在15个观察到的MAG中,有10个至少包含一个与BA生成有关的基因(每个MAG中包含一个到五个基因)。MAG中的这些基因参与了四种类型的BA合成,包括腐胺(speA、speB、aguA、arg、odc和ptcA)、尸胺(ldc)、精胺(speE)和酪胺(tyrDC)(图5B),反映了酱油发酵过程中不同物种或菌株的BA生产能力。在检测到的四个脱羧酶基因(speA、odc、ldc和tyrDC)中,没有发现所有MAG中都有一个单一的基因。在腐胺的产生方面,7个属于芽孢杆菌属(MAG14)、棒状杆菌属(MAG12)、克雷伯氏菌属(MAG13)、片球菌(MAG15)、罗斯氏菌属(MAG1)、葡萄球菌属(MAG8)和动物球菌属(MAG9)的MAG携带了speB基因(图5B)。除了两个MAGs(MAG13和MAG15)外,上述所有MAG都携带arg基因。K. pneumoniae(MAG13)和P. pentosaceus(MAG15)被发现拥有odc基因。在归类为K. pneumoniae(MAG13)和P. acidilactici(MAG4)的MAG中鉴定出了speA、aguA和ptcA基因。这些结果表明,在酱油发酵过程中,有可能通过两种不同的途径从精氨酸合成腐胺:鸟氨酸脱羧途径(ODC)和肌氨酸脱氨基途径(AGDI)。ODC途径涉及将精氨酸转化为鸟氨酸(arg),然后使鸟氨酸脱羧形成腐胺(odc),而AGDI途径涉及将精氨酸脱羧形成肌氨酸(精氨酸脱羧酶,speA),使肌氨酸脱氨基形成N-氨甲酰腐胺(肌氨酸脱氨基酶,aguA),最后使N-氨甲酰腐胺水解形成腐胺(腐胺氨甲酰转移酶,ptcA)。令人意外的是,这些MAG中没有一个包含了上文提到的腐胺完整生产途径,因为它们并不包含与这一特定途径相关的所有基因,这表明在腐胺的生产过程中,不同的MAG之间存在协同作用。这在一定程度上与之前的报告相符,那些拥有ODC途径以生产腐胺的物种被检测到,而拥有完整的AGDI途径的物种则未被发现。考虑到K. pneumoniae(MAG13)拥有两个基因(speA和speB),推测这些物种可能通过精氨酸的脱羧反应(speA)和肌氨酸的酶解作用(speB)来产生腐胺。已有报告表明,K. pneumoniae能够产生腐胺、组胺和尸胺。此外,ldc基因仅在隶属于K. pneumoniae的MAG13中被发现。有趣的是,在所检测的所有菌种中,只有K. pneumoniae携带了超过两个的脱羧酶基因(speA、odc和ldc)(图5B),这表明该菌可能有能力脱羧多种氨基酸(精氨酸、鸟氨酸和赖氨酸)。此外,两个MAG菌株携带了编码脱羧酶的tyrDC基因,包括屎肠球菌(MAG10)和类肠膜魏斯氏菌(MAG5),这表明它们可能通过从各自的预氨基酸酪氨酸进行一步脱羧反应来产生酪胺。屎肠球菌经常在各类发酵食品中被分离或检测到。本研究中关于屎肠球菌菌种酪胺产生的研究结果并不出人意料,因为从各种发酵食品中分离出的屎肠球菌菌株大多已被鉴定为酪胺和苯乙胺产生菌,且已被发现携带tyrDC基因。据报道,从韩国泡菜中分离出的类肠膜魏斯氏菌菌株能够产生多种胺类物质,包括酪胺。尤其值得注意的是,在T. halophilus(MAG2)中并未发现BA基因,而在JP和CP发酵的中后期阶段,T. halophilus是最占优势的菌种(图5A)。这一发现与之前的报告相似,即在14个T. halophilus菌株中,只有一个菌株的基因组中鉴定出了单一的组氨酸脱羧酶基因。但与多项研究结果并不一致,这些研究显示某些T. halophilus菌株携带了多种BA基因,如酪氨酸和组氨酸脱羧酶基因,在发酵食品中发挥着积累酪胺和组胺的关键作用。此外,在微生物群落水平上检测到的参与组胺(hdc)和色胺(tdc)合成的基因(图3)并未在任何一个MAG中被识别,这表明这些基因可能存在于本研究中的其他未组装微生物中,因此需要更深入的宏基因组测序。最后,关于这些关键物种(MAGs)在酱油发酵过程中产生BA的预测代谢途径概览展示在图5C中。鉴于鸡葡萄球菌(MAG8)在CP发酵的中后期阶段具有较高的丰度(图5A),且其携带有产生腐胺的基因(arg和speB)(图5C),推断腐胺的高含量与这一物种在CP样本中的高丰度有关。
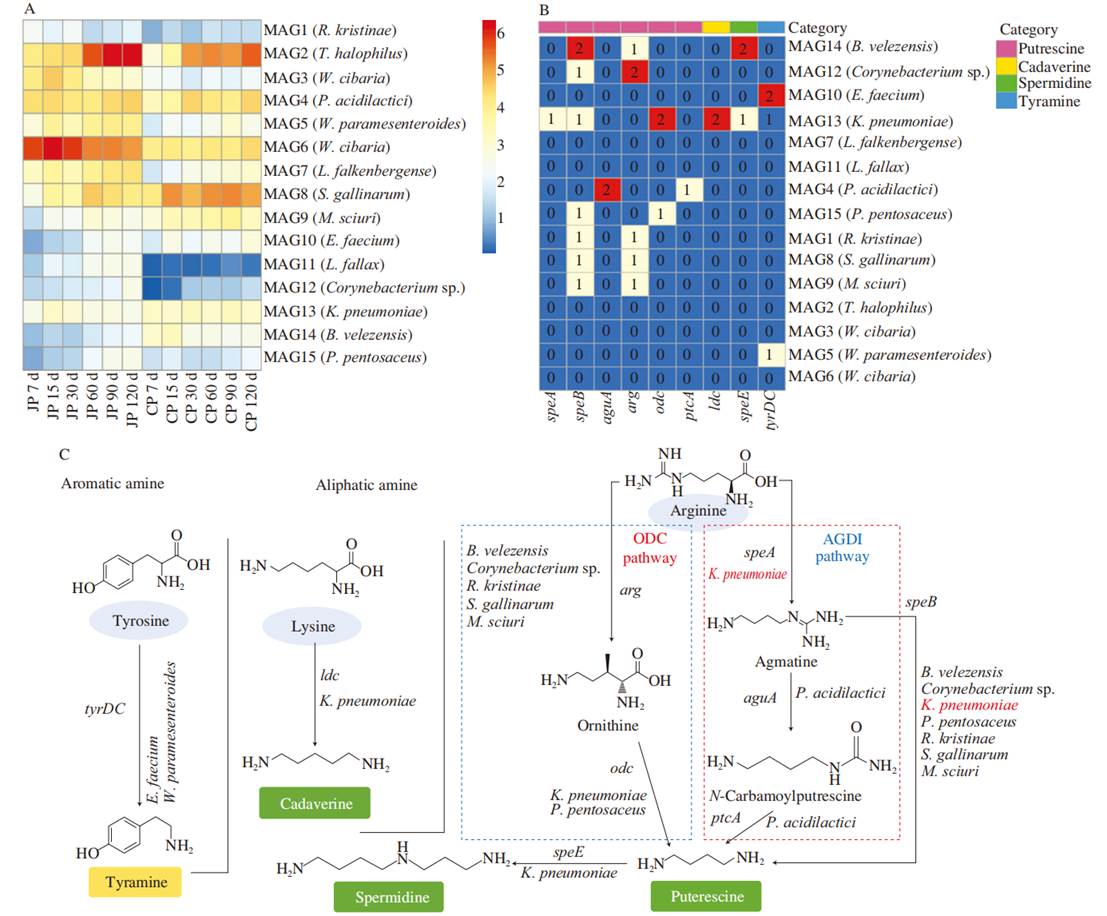
图5(A)各混样MAG的丰度分布,(B)MAG中的BA基因,(C)预测的大酱发酵过程中参与BA形成的MAG
四、研究结论
这项研究确定了BA谱型和微生物群落组成上的差异,并观察了两种不同类型的酱油发酵过程(JP和CP)中负责BA生成的微生物以及相关代谢途径。CP样品中三种BA(腐胺、酪胺和组胺)的含量显著高于JP样品。最丰富的细菌属包括四联球菌属、葡萄球菌属和芽孢杆菌,但其相对丰度在不同发酵类型间存在差异。对微生物群落和功能基因组进行注释,揭示了与BA合成相关的微生物功能潜力。特别是,从宏基因组学数据中恢复的微生物基因组进一步细化了关键成员的了解,并揭示了与酱油发酵过程中涉及的各种BA生成基因相关的发现。未来需要进一步努力,通过宏转录组学评估基因表达,并研究不同物种在酱油发酵过程中的代谢相互作用。
参考文献
Tan G, Wang Y, Hu M, et al. Insights into the biogenic amine-generating microbes during two different types of soy sauce fermentation as revealed by metagenome-assembled genomes. Food Science and Human Wellness, 2025, 14(3): 9250064. https://doi.org/10.26599/FSHW.2024.9250064
密码子云平台
我们推出的密码子生信云平台服务(https://cloud.mimazi.net),包含细菌基因组云流程和各种生信分析免费小工具,无需安装软件、无需配置环境,即可一键化生成数据分析及可视化绘图结果,快来试试吧!
-
点赞 (0人)
- 收藏 (0人)
-
上一篇: 没有了
下一篇: 没有了



