掌握数据可视化利器:Upset图(集合图)绘制方法详解
- 看不见的线
- 7297
- 2024-05-06 09:47:46
- 原创
在数据分析和可视化中,经常需要对某几组样本中共有或特有的基因,OTU或微生物进行可视化,通常可以选择维恩图进行可视化。但当分组信息过多,维恩图的展示能力及可读性则有所下降,因此推荐使用维恩图的升级版本—集合图(Upset plot)。

集合图主要由三部分组成
最上面的y轴以柱状图形式展示,用于表示交集大小,
下面左侧的x轴以条形图形式展示,表示每一个集合的大小,
下面右侧的矩阵点表明集合之间的交集情况,例如,若两个样本间有相同的元素,则使用连线将两个样本相连;同样地,若多个样本间存在连线,那这部分即为这几个样本内的共有元素。
下面让我们一起学习如何使用R语言绘制集合图。
示例数据(data1.csv)
数据为丰度/表达量矩阵,表格需要带表头和列名,每一列为样本名,每一行为各种指标数据名,如OTU,基因ID,代谢物名称等。

集合图绘制示例
#读取本地数据并查看
dat <- read.csv("data1.csv", header = TRUE,row.names = 1,check.names = F)
head(dat)

#加载R包
# install.packages("UpSetR")
library(UpSetR)
#将丰度矩阵中的非0值转换为1
dat[dat > 0] <- 1

#集合图绘制
#基础绘图
upset(dat)
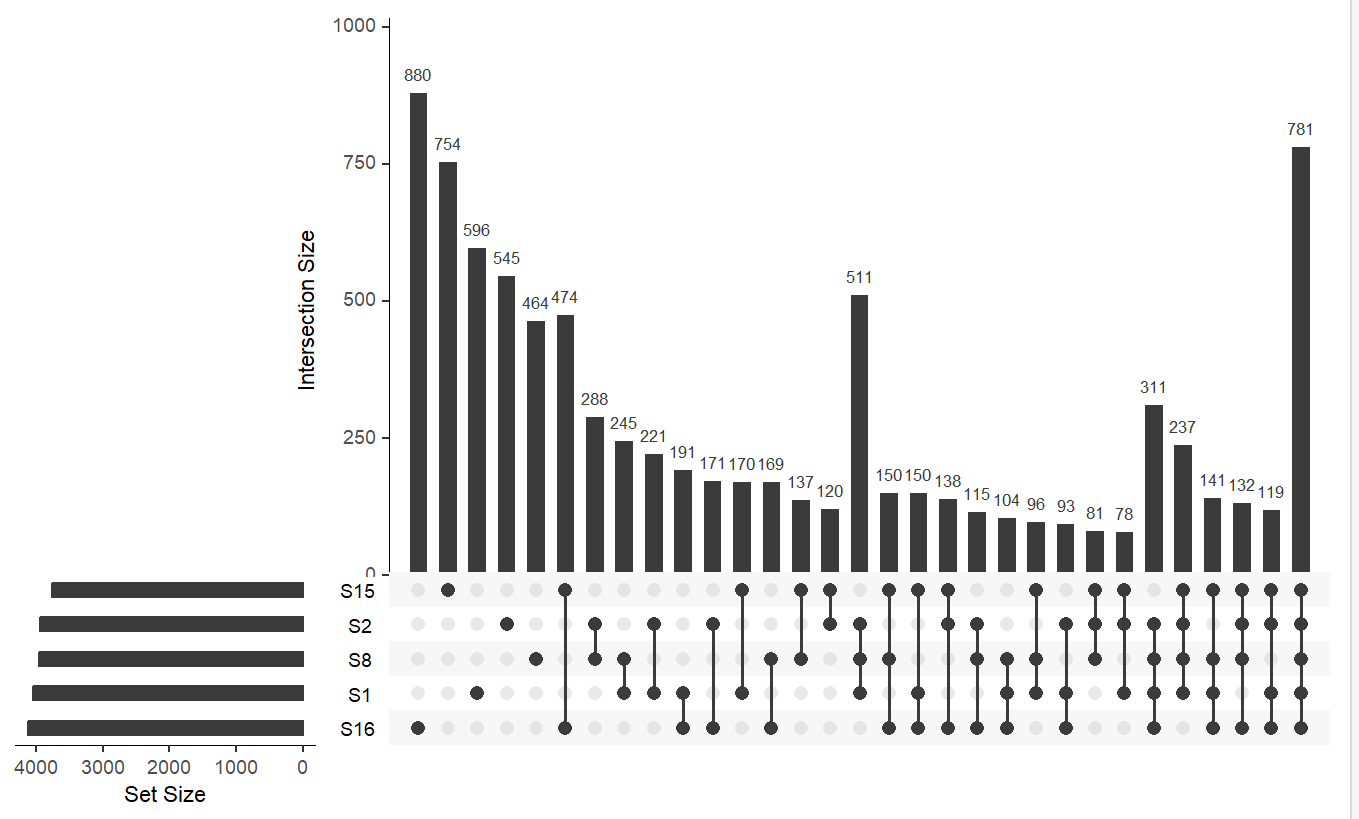
#集合图美化
upset(dat,
nsets = 20, #可视化数据集数量
nintersects= 60, #显示前多少个交集
main.bar.color = 'blue', #柱状图颜色
matrix.color="black", #集合点的颜色
sets.bar.color= "red", #条形图条形的颜色
set_size.show = F, #是否在条形图上显示集合大小
mb.ratio = c(0.5, 0.5) #矩阵图与主柱图之比
)
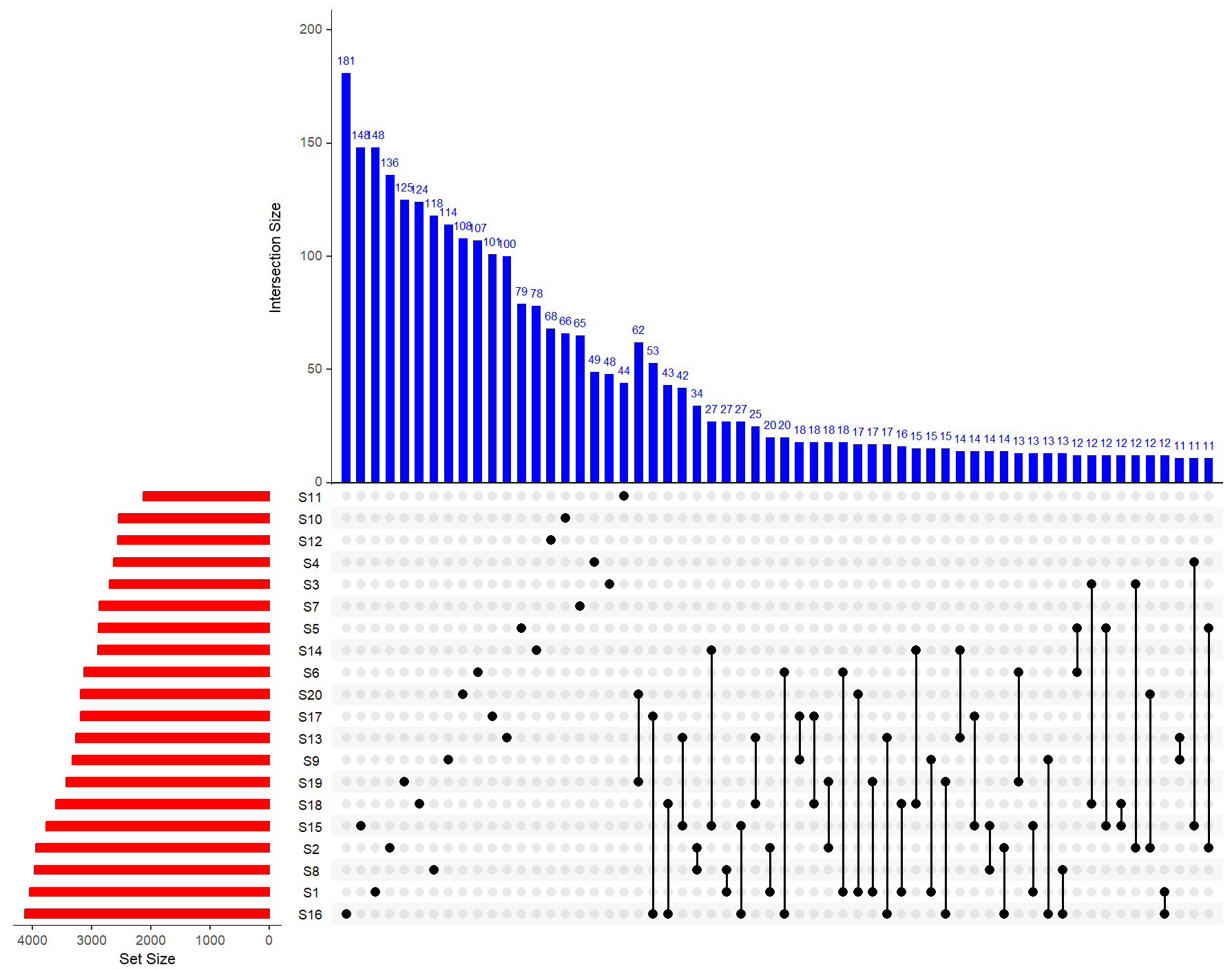
#将条形图颜色设置为不同的颜色
# install.packages("RColorBrewer")
library(RColorBrewer)
upset(dat,
nsets = 20, #可视化数据集数量
nintersects= 60, #显示前多少个交集
main.bar.color = 'blue', #柱状图颜色
matrix.color="black", #集合点的颜色
sets.bar.color= c(brewer.pal(10,"Set3"),brewer.pal(10,"Paired")), #条形图条形的颜色
set_size.show = F, #是否在条形图上显示集合大小
mb.ratio = c(0.5, 0.5) #矩阵图与主柱图之比
)
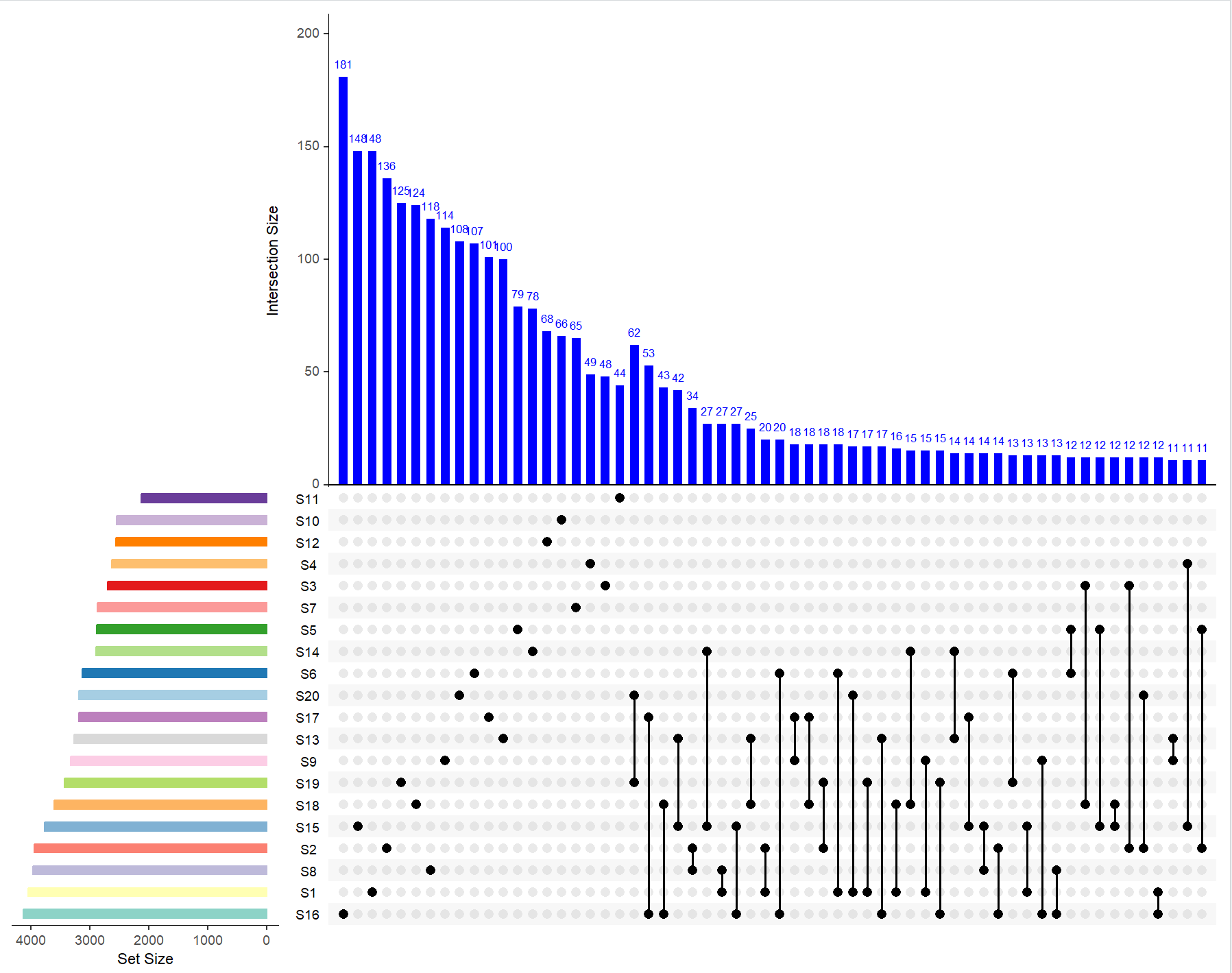
#高亮显示特定交集
# queries参数用于指定要显示的特定组的交集,每个元素都包含一个query(查询类型)和params(查询参数)字段。在这个例子中,有两个查询:第一个查询指定"S1"和"S8"集合的交集,将其显示为红色;第二个查询指定"S15"集合,将其显示为绿色。color字段用于设置交集的颜色,active字段用于指定是否激活该查询
upset(dat, nsets = 20,mb.ratio = c(0.5, 0.5),
#设置自己想要展示的特定组的交集
queries=list(list(query=intersects, params=list("S1","S8"),
color="red",active=T),
list(query=intersects, params=list("S15"),
color="green",active=T))
)
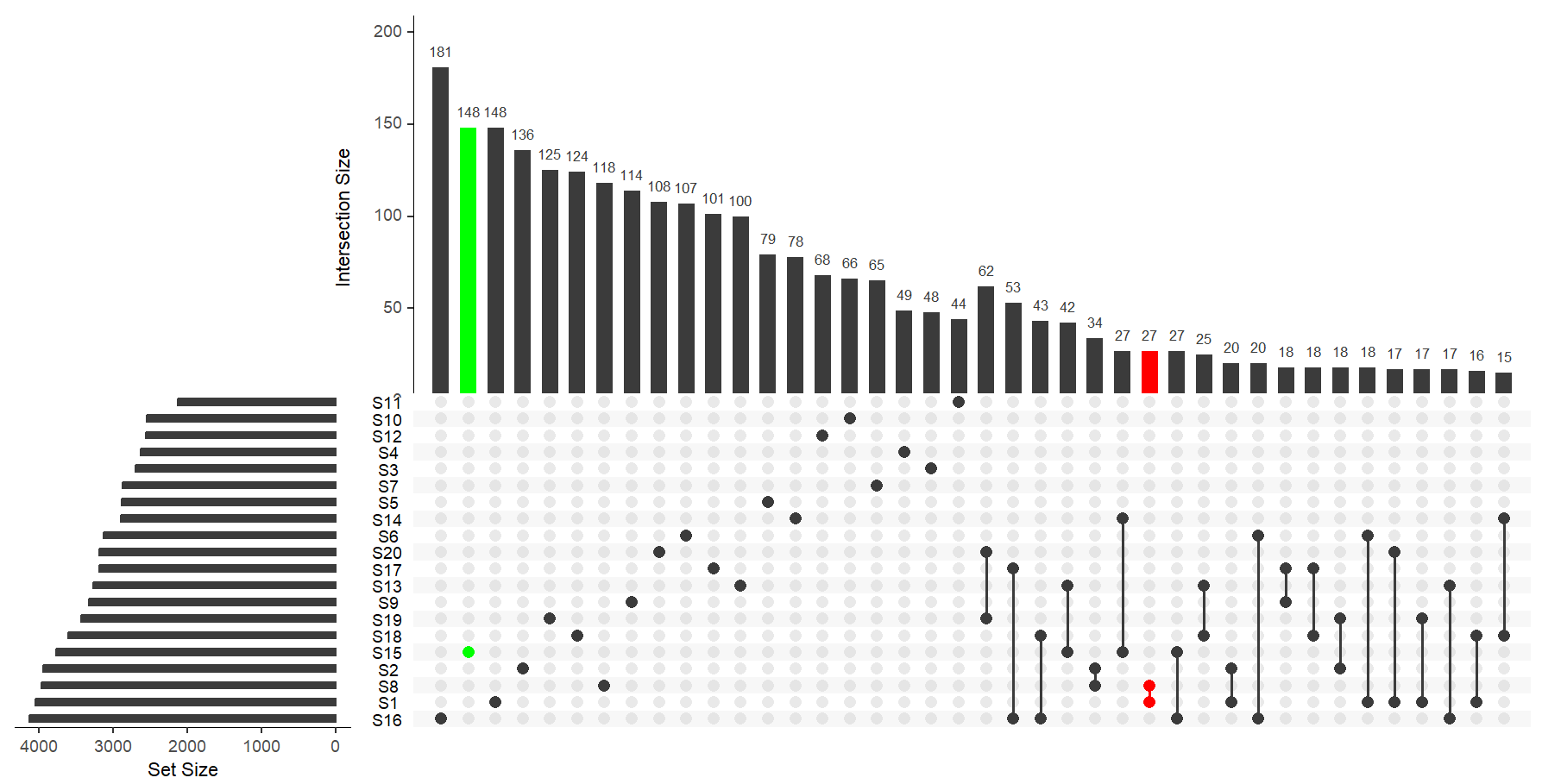
推荐课程
【课程】微生物比较基因组精品系列课——全套自学必入的系统课程
课程链接:微生物比较基因组精品系列课【全套】
【课程】铜绿假单胞菌基因组研究和分子分型实战
课程链接:铜绿假单胞菌基因组研究和分子分型实战
【课程】微生物比较基因组与群体进化——基因组变异专题研究
课程链接:微生物比较基因组与群体进化
【课程】微生物分子分型-MLST课程——分型全套(含理论、软件、方法)
课程链接:微生物分子分型-MLST课程
【课程】基因组结构分析神器Easyfig实操精品课
【课程】BRIG绘图——结构比较专题2
【课程】肺炎克雷伯菌基因组学研究综合指南
课程链接:肺炎克雷伯菌基因组学研究综合指南
【课程】微生物基因组生信必学课程
课程链接:微生物基因组生信分析必学课程
【课程】微生物生防菌研究
课程链接:生防菌的系统化研究
专题材料
【资料】耐药专题材料
【资料】生防专题材料
请添加唯那生物技术客服小唯的微信号winnerbio01,备注“耐药专题”或“生防专题”,立马获取。
更多专题推荐
1、生信技能课专辑
2、耐药毒力专题
3、肺炎克雷伯菌专题
4、密码子偏好性专题
7、软件脚本分享
9、知识干货
10、分泌系统
11、软件下载
12、书籍下载
-
点赞 (0人)
- 收藏 (0人)



