探索数据关系,解读趋势变化——用R语言轻松进行相关性分析
- 看不见的线
- 5295
- 2024-06-03 17:41:50
- 原创
在进行数据分析时,相关性分析是一项重要的技术,可帮助我们理解数据之间的关系,揭示变量之间的联系。而R语言作为一款强大的统计分析工具,提供了丰富的函数和包,便捷地进行相关性分析。今天我们为大家介绍如何使用r语言进行相关性分析。
相关性分析的意义与应用场景
相关性分析用于评估两个或多个变量之间的关联,能通过定量指标描述变量之间的强弱、直接或间接联系。
相关系数
是对变量之间的相关程度的定量描述
- 相关系数值介于-1~1之间
- 越接近0相关性越低,越接近-1或1相关性越高
- 正负号表明相关方向,正号为正相关、负号为负相关
常见相关系数
| 系数 | 数据类型 | 假设前提 | 鲁棒性 |
| Pearson | 适用于连续变量,要求数据呈现线性关系 | 数据呈正态分布,要求线性相关性假设 | 对异常值敏感,受极端值影响较大 |
| Kendall | 适用于顺序数据,不要求数据呈现线性关系 | 无需假设数据分布,适用于非线性相关性 | 对异常值不敏感,稳健性较好 |
| Spearman | 适用于顺序数据和连续变量,不要求数据呈现线性关系 | 无需假设数据分布,适用于非线性相关性 | 对异常值不敏感,稳健性较好 |
相关性分析应用场景
组内相关性分析,如评估多个样本/基因/功能通路间的相关性
组间相关性分析,如物种与功能基因,代谢通路的关联,环境因子与基因表达的关联等
示例数据:
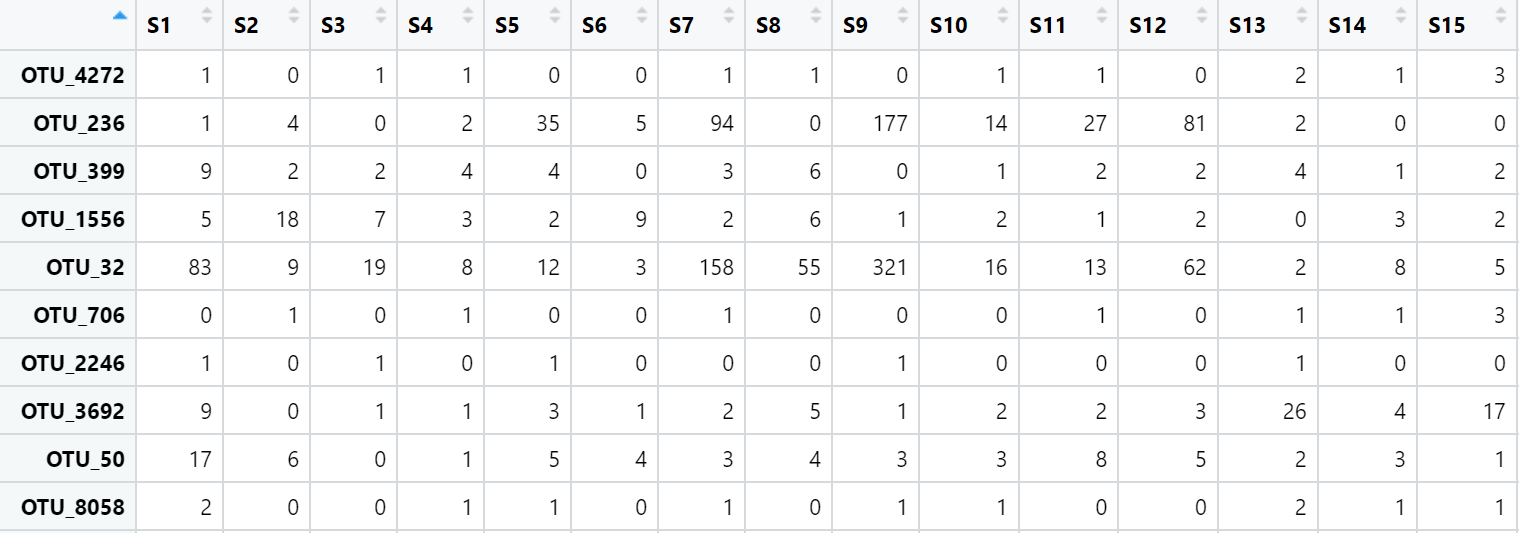
otu丰度表(otu_filtered.csv)
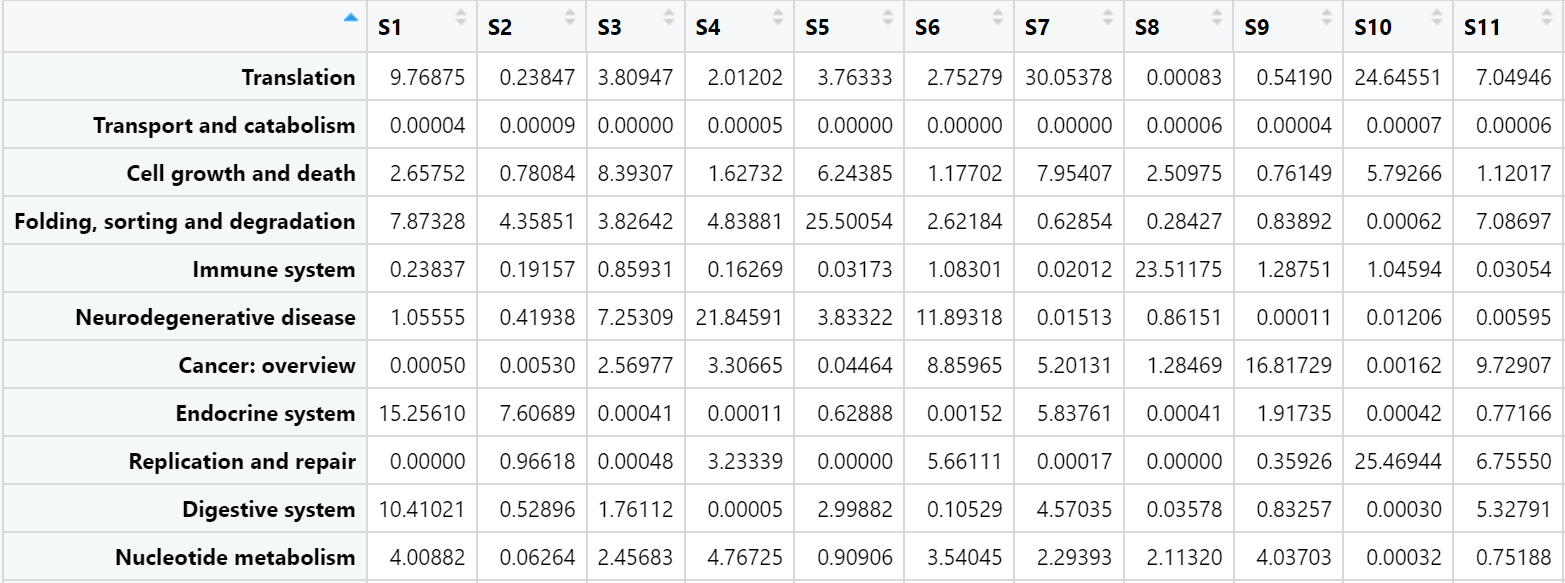
功能通路丰度表(fun.csv)
#使用R语言进行相关性分析
#清空环境变量
rm(list = ls())
#设置工作目录
setwd("D:/r_class/test_data/4.cor_analysis")
#进行相关性分析
#安装和加载R包
# install.packages("psych")
library(psych)
# 1、组内相关性分析
#以otu丰度矩阵为例,比较样本间的相关性
#读取本地数据
#读取otu丰度矩阵
otu <- read.csv("otu_filtered.csv",header = T,
row.names = 1,check.names = F)
head(otu)

#正态分布检验,确定选择哪种相关性分析方法(满足正态分布使用Pearson相关系数,不满足正态分布使用Spearman相关系数)
# shapiro.test()函数:检测3~5000个数值向量是否符合正态分布
#如果 p值小于 0.05,则有足够的证据表明样本数据分布不满足正态分布
shapiro.test(otu$S1)

#执行相关性分析
#函数帮助信息查看
?corr.test
#样本间的相关性
# method参数可选:"spearman" , "kendall", "pearson"
# adjust参数可选:"holm", "hochberg", "hommel", "bonferroni", "BH", "BY", "fdr", "none"
cor_sample <- corr.test(otu,
method = "spearman" , #相关性分析方法
adjust= "fdr" #多重检验校正方法
)
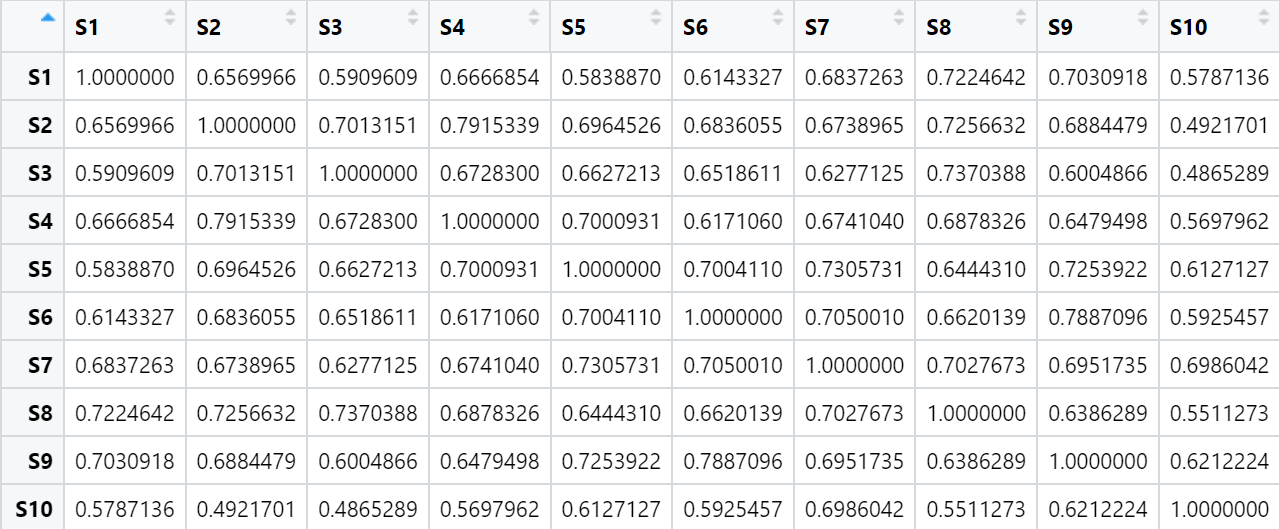
#提取相关性、p值
#提取相关性矩阵
cmt <- cor_sample$r
#提取丰度矩阵
pmt <- cor_sample$p
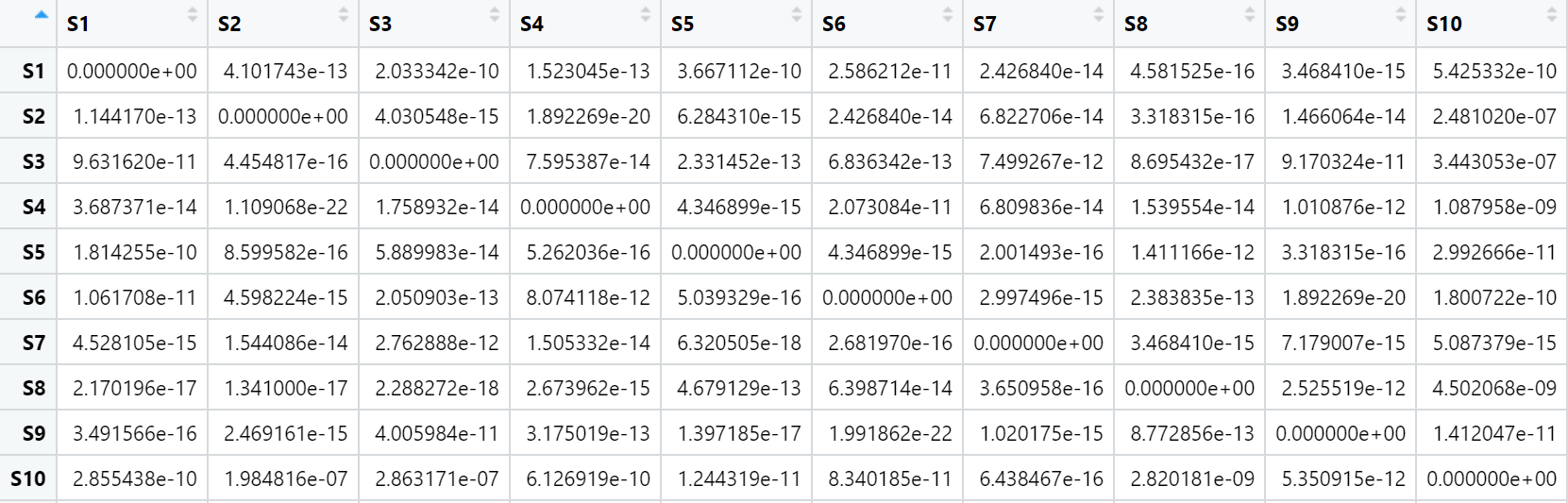
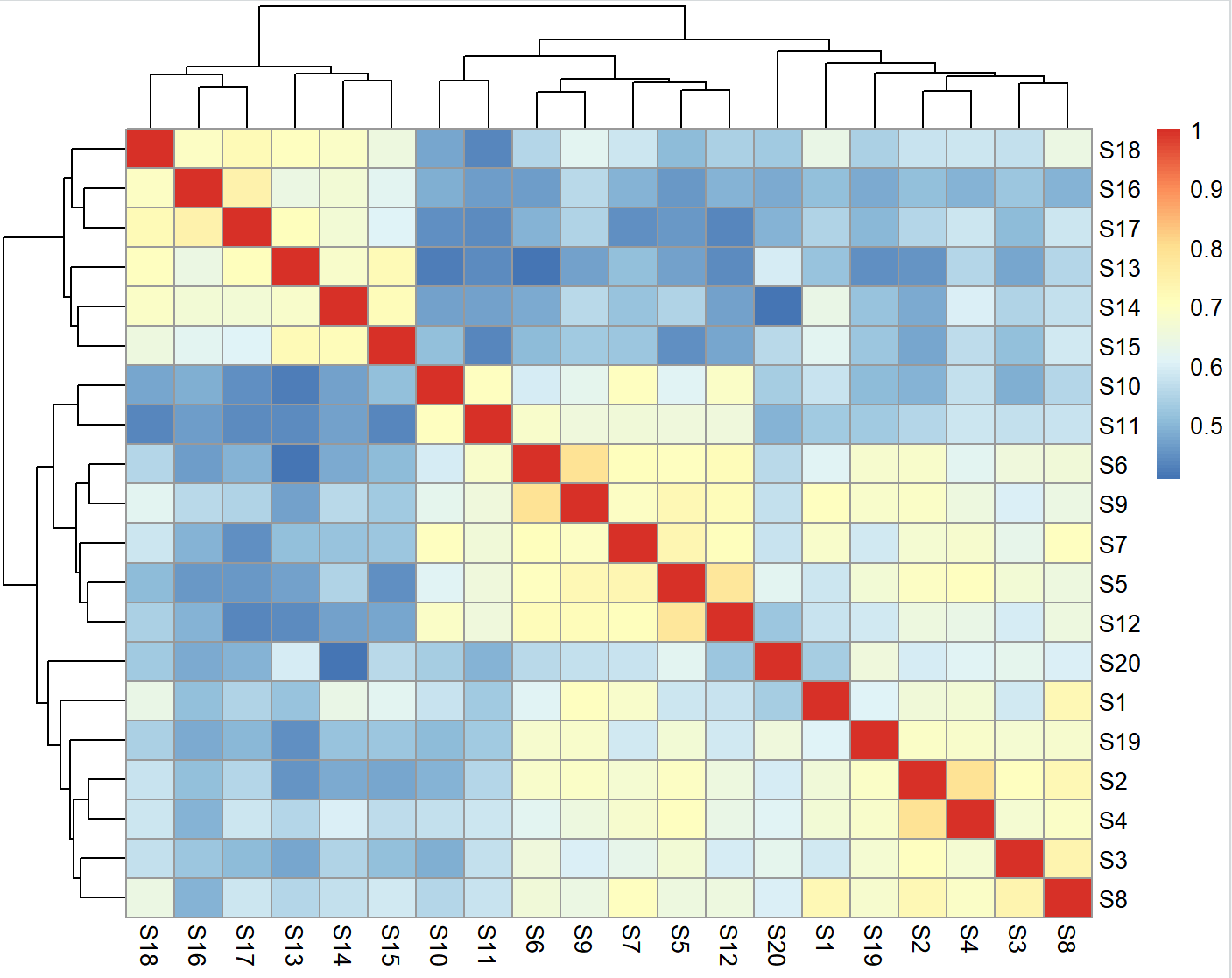
#绘制热图展示样本间的相关性
# installed.packages("pheatmap")
library(pheatmap)
pheatmap(cmt)
# 2、组间相关性分析
#以otu丰度矩阵和功能注释矩阵为例,比较otu与功能间的相关性
#读取本地数据
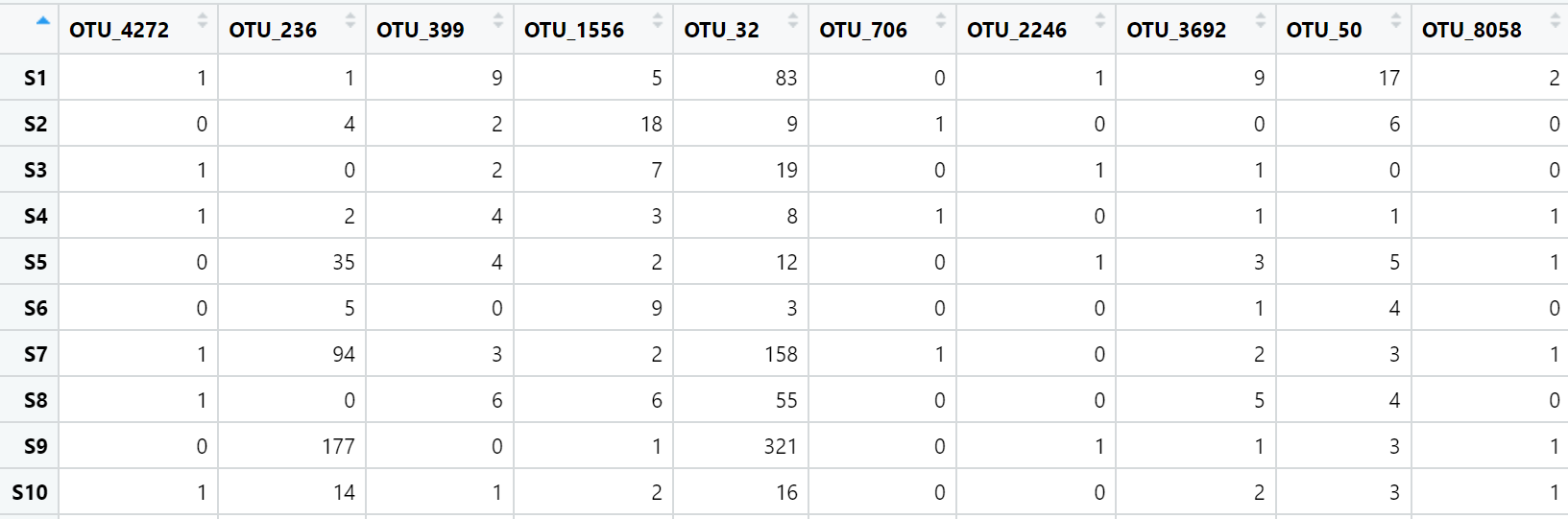
#读取otu丰度矩阵
otu <- read.csv("otu_filtered.csv",header = T,
row.names = 1,check.names = F)
head(otu)
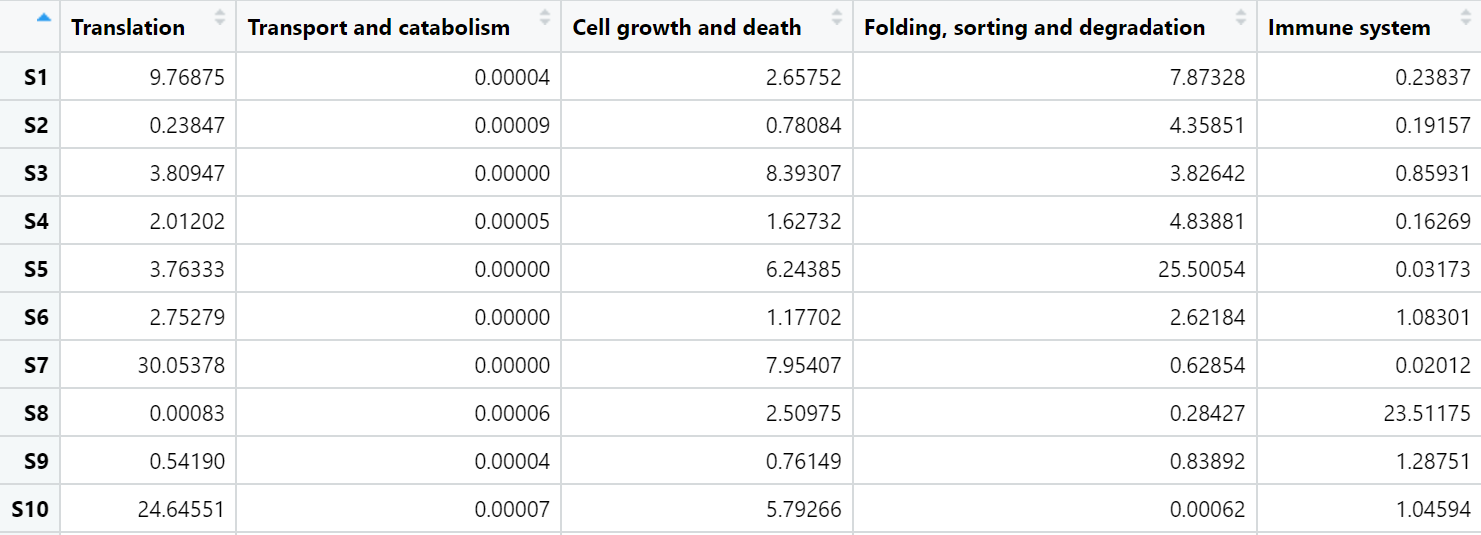
#读取功能注释结果
fun <- read.csv("fun.csv",header = T,
row.names = 1,check.names = F)
head(fun)
#行列转换,每行代表一个样本,每列代表一个otu或功能
otu_t <- t(otu)
fun_t <- t(fun)
#执行组间相关性分析
cor <- corr.test(otu_t, fun_t,
method = "pearson" , #相关性分析方法
adjust= "fdr" #多重检验校正方法
)
#提取相关性、p值
cmt1 <-cor$r
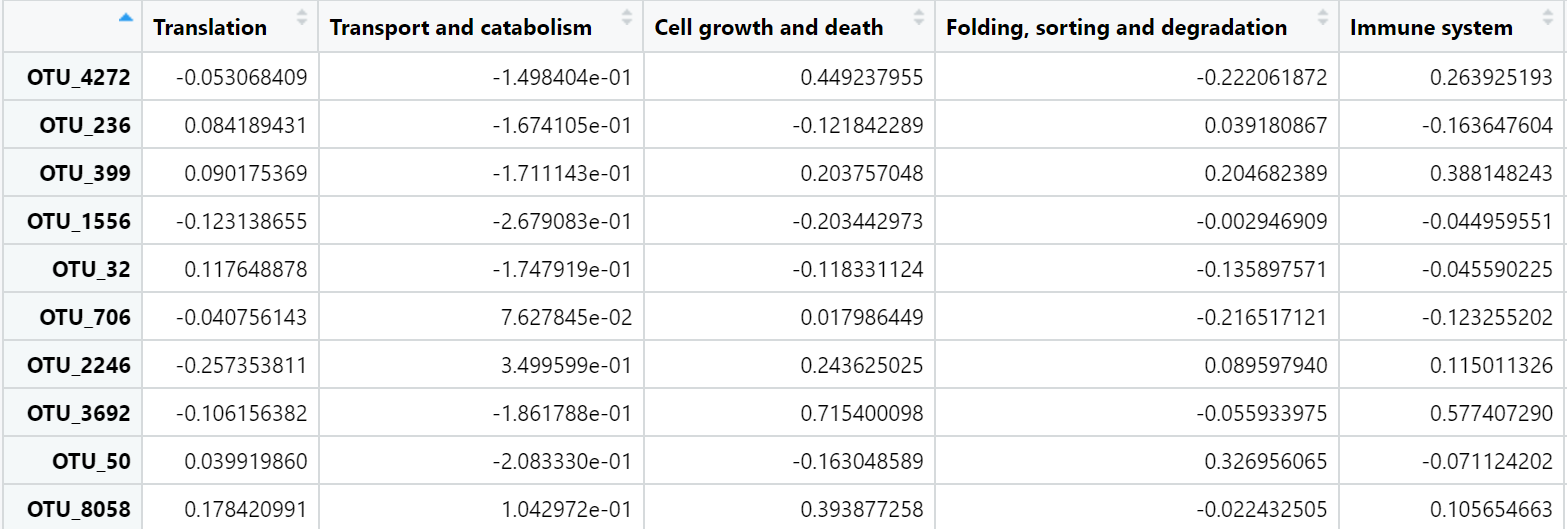
pmt1 <- cor$p
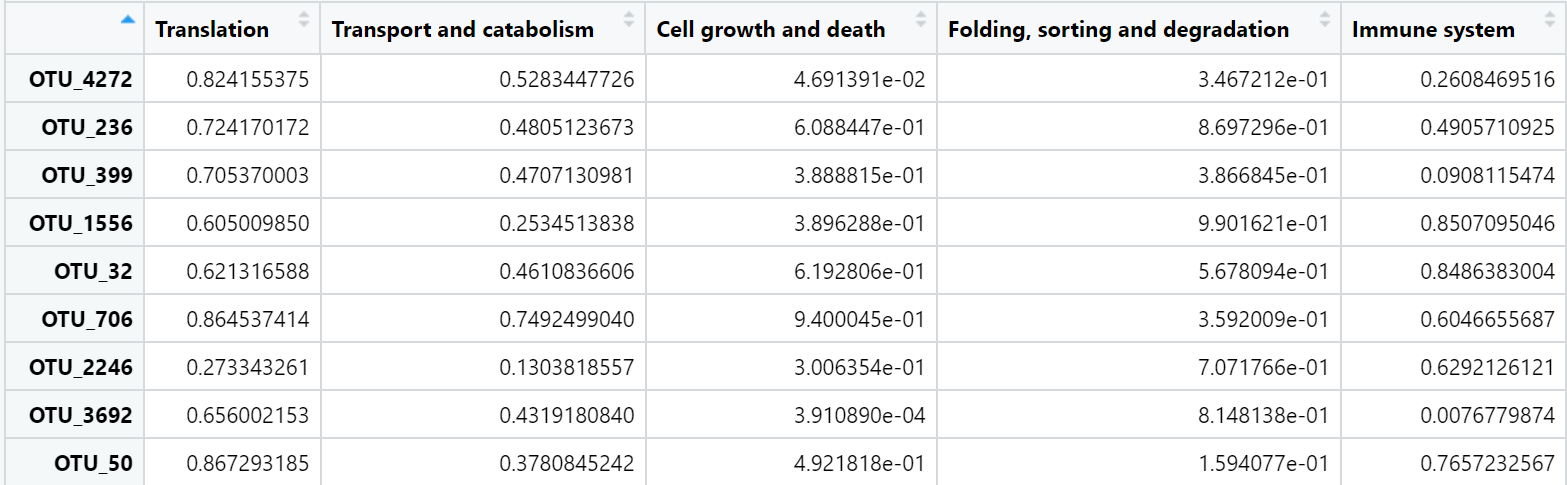
#绘制热图展示otu与功能间的相关性
pheatmap(cmt1,angle_col = 45)

推荐课程
【课程】R语言入门与高通量测序数据实战处理
课程链接:R语言入门与高通量测序数据实战处理
【课程】微生物比较基因组精品系列课——全套自学必入的系统课程
课程链接:微生物比较基因组精品系列课【全套】
【课程】铜绿假单胞菌基因组研究和分子分型实战
课程链接:铜绿假单胞菌基因组研究和分子分型实战
【课程】微生物比较基因组与群体进化——基因组变异专题研究
课程链接:微生物比较基因组与群体进化
【课程】微生物分子分型-MLST课程——分型全套(含理论、软件、方法)
课程链接:微生物分子分型-MLST课程
【课程】基因组结构分析神器Easyfig实操精品课
【课程】BRIG绘图——结构比较专题2
【课程】肺炎克雷伯菌基因组学研究综合指南
课程链接:肺炎克雷伯菌基因组学研究综合指南
【课程】微生物基因组生信必学课程
课程链接:微生物基因组生信分析必学课程
【课程】微生物生防菌研究
课程链接:生防菌的系统化研究
专题材料
【资料】耐药专题材料
【资料】生防专题材料
请添加唯那生物技术客服小唯的微信号winnerbio01,备注“耐药专题”或“生防专题”,立马获取。
更多专题推荐
1、生信技能课专辑
2、耐药毒力专题
3、肺炎克雷伯菌专题
4、密码子偏好性专题
7、软件脚本分享
9、知识干货
10、分泌系统
11、软件下载
12、书籍下载
-
点赞 (0人)
- 收藏 (0人)
-
上一篇: 没有了
- 下一篇: R与RStudio的详细安装教程



