Nucleic Acids Research| oriTfinder: 细菌耐药质粒接合转移研究工具
- 唯那生物
- 660
- 2023-01-06 14:12:57
- 原创
oriTfinder: a web-based tool for the identification of origin of transfers in DNA sequences of bacterial mobile genetic elements
《《《导读》》》
由于抗菌药物的不合理使用,病原细菌的耐药性日趋加剧。细菌可移动遗传元件(mobile genetic elements, MGE),如接合质粒和整合性接合元件(Integrative and conjugative element, ICE),是细菌致病性和耐药性传播的重要载体。接合性质粒和ICE的接合转移区通常由四个模块组成:转移起点(oriT)区,松弛酶 (relaxase),IV型伴侣蛋白 (type IV coupling proteins, T4CP)和细菌IV型分泌系统(type IV secretion system, T4SS),展现出滚环复制和接合转移等典型的单链DNA自行转移特性。在接合转移过程中,松弛酶识单链DNA(ssDNA)转移起始位点(oriT),结合并切割,被切割的单链进行滚环复制,T4CP识别并与ssDNA结合,随后通过T4SS从供体细胞转移到受体细胞中。相对于接合性可移动元件,大量非接合(non-conjugative) 性可移动元件(MGE),包括可移动质粒和整合性可移动元件(integrative and mobilizable elements,IMEs),可以进行移动,但是不能进行自主传播,基因组上通常携带有限数量的mob基因用于其在接合中的自身DNA加工。有趣的是,携带功能性oriT序列的非接合MGE可以通过接合元件进行传播。例如,携带oriT的霍乱弧菌基因组岛可借由SXT/R391 ICE进行传播。SXT元件还可以介导质粒RSF1010的传播,该质粒编码对磺酰胺和链霉素的抗性。oriT区域的长度通常为数十至数百bp,包含保守的切口区域(位于nic位点的侧翼)和可变数目的反向重复序列(IR)。nic位点被松弛酶识别和切割,而IR参与松弛酶对nic位点的精确识别以及ssDNA转移的终止。因此,鉴定MGE序列中的oriT区域对于研究MGE的自主转移能力(self-transfer or mobilizing transfer capability)是非常重要的。
————
oriTfinder在线工具的开发:
截至目前,已有多种用于预测MGE T4SS的生物信息学工具,如SecReT4数据库,AtlasT4SS数据库,EffectiveDB数据库,基于web的T346Hunter和在线工具VRprofile。然而,这些数据库中都缺乏ssDNA转移起始相关的数据,包括oriT区域,松弛酶和T4CP。
实际案例:
肺炎克雷伯菌是一种重要的病原菌,是世界范围内造成医院感染的重要病原体。它目前被认为是全球主要的抗生素耐药性来源和传播工具。在本项研究中,研究团队从GenBank中截至2017年12月1日的107个完全测序的肺炎克雷伯菌基因组中收集了311个质粒序列。这些质粒的不相容性由PlasmidFinder确定。使用oriTfinder,成功鉴定了这些质粒的转移区,包括oriT,松弛酶,T4SS和T4CP(Fig.1)。在311个质粒中,发现26.4%(82/311)具有一整套oriT,松弛酶,T4cP和T4SS,表明它们具有较强的自我转移能力。值得注意的是,在这82个接合质粒中,其中有63个质粒携带抗生素抗性基因(AR),表明这些质粒可能介导了抗生素抗性基因传播。例如,在分离自临床的肺炎克雷伯菌HS11286的111kb碳青霉烯酶编码(carbapenemase-encoded)的质粒pKPHS2中含有与oriTDB数据库中质粒R100同源的oriT区,其H值为0.51。此外,发现有12.2%(38/311)的质粒具有oriT和松弛酶,但缺乏T4CPs和/或T4SS,表明它们是潜在的可移动质粒,在这38个可移动质粒中,有31个携带获得性抗生素抗性基因,其可以通过接合性MGE进行传播。另外,61.4%(191/311)质粒未预测到相关的接合转移模块,而在这191个不可转移质粒中,有20个携带AR基因。
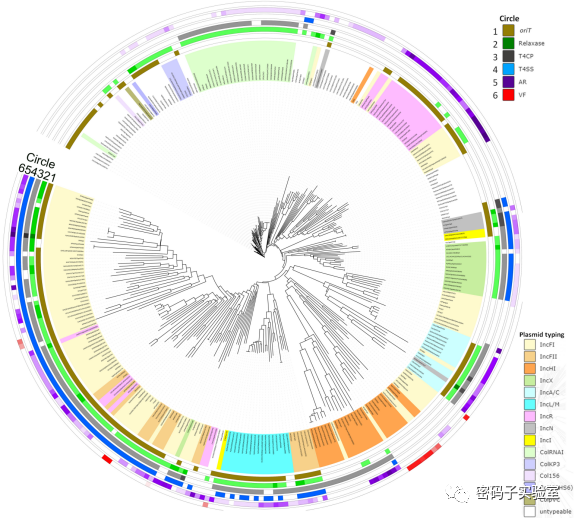
Figure1.An overview of the oriTfinder-facilitated detection of putativeoriTregions and three other conjugal modules in the complete sequences of 311 plasmids of the 107 completely sequencedK. pneumoniaegenomes.The phylogenetic patterns based on the presence/absence of orthologous gene families of all the plasmids under analysis. A binary gene presence/absence matrix was created using OrthoMCL with defaultsettings(4)and a hierarchical cluster result was shown by iTOL(5). The plasmids belonging to the reported incompatibility groups are marked in different colours. The six colour circles denote the regions (or genes) present in a given plasmid, includingoriT, relaxase, T4CP, T4SS, AR gene and VF gene from the inside out. The gradient of colour of each circle represents the variable numbers of genes or gene clusters.
上海唯那生物专注于提供微生物包括耐药性研究在内的各类个性化服务,包括基金方案申报、组学测序、个性化生信分析、文章辅导发表、科研绘图、微生物纯化培养、基因克隆、微生物突变体(库)构建等。全方位,一站式,我们期待与您的合作。从数据处理、相关运算,到结果解读、图片绘制,全方位、一站式提供相关研究的解决方案,大家有需要的话也请联系我们。
>>
唯那生物精心准备的微生物耐药专题解析合集材料,具体包括:
1、微生物耐药研究快速入门综述;
2、微生物耐药研究经典案例解读;
3、微生物耐药研究专题方案书;
4、微生物耐药研究的热点好文章。

-
点赞 (0人)
- 收藏 (0人)



