微生物基因组研究文章解读l 1
- 唯那生物
- 406
- 2023-01-09 17:44:11
- 原创
这是一个关于微生物基因组研究的系列内容,旨在为初步涉入基因组学研究的同学查缺补漏,答疑解惑。本系列内容涉及微生物基因组学的研究方法、研究思路导读以及最新研究进展,希望对大家有用。大家有其他相关的问题,可以在后台留言,我们会尽力在下期为您解答。
耐药基因近点案例1
Phylogeneticallyinformative mutations in genes implicated in antibiotic resistancein Mycobacteriumtuberculosis complex
作者:MatthiasMerker等
期刊:GenomeMed
时间:2020.3.6
影响因子:10.675
DOI:10.1186/ s13073-020-00726-5
主要方法:Illumina技术(MiSeq,NextSeq500,HiSeq2500);耐药基因SNP系统发育树分析;MIC耐药性检测
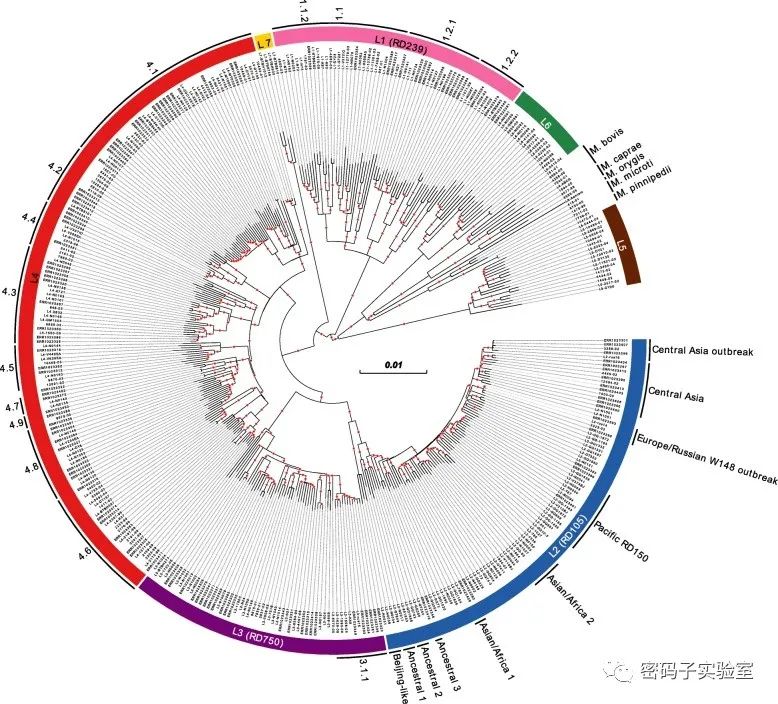
研究思路:为了准确地解释基因型药物敏感试验(drugsusceptibility testing,DST)的全基因组测序数据,需要全面了解结核分枝杆菌复合物(Mycobacteriumtuberculosis complex,MTBC)中与抗生素耐药相关的基因的遗传变异的关系。该作者对405个MTBC菌株进行了21种抗结核药物耐药性的92个基因的SNP检测,如下图所示分为了7个进化分支,确定基因型与表型之间的关系。
耐药基因近点案例2
Convergenceof virulence and MDR in a single plasmid vector in MDR Klebsiellapneumoniae ST15
作者:MargaretM C Lam等
期刊:JAntimicrob Chemother
时间:2019.2.15
影响因子:5.113
DOI:10.1093/ jac / dkz028
高通量测序方法:使用Illumina和Oxford纳米孔测序法解析了两个分离物的完整基因组序列,包括它们的质粒。
研究思路:分别在IlluminaMiSeq和HiSeq平台上针对12个ST15 Kp分离株测序。为了解析菌株的完整质粒序列,在MinION(OxfordNanoporeTechnologies)上进行长读长测序,并与Illumina的短读物结合以产生杂合体。比较基因组分析:2003至2015年从七家医院分离出的10个ST15菌株、报告KpST15基因组序列的论文中鉴定出的公开数据、EuSCAPE欧洲公司调查产生碳青霉烯酶的肠杆菌科收集的Kp基因组数据鉴定ST15分离株,用于比较基因组分析。将所有序列定位到KP_104014的基因组,推断核心基因组最大似然系统发育。从定位数据还得到pKp104014_1序列的覆盖范围,KP_104014的hv-MDR质粒的覆盖范围以及在pKp104014_1中注释的基因。
文章主要结果:
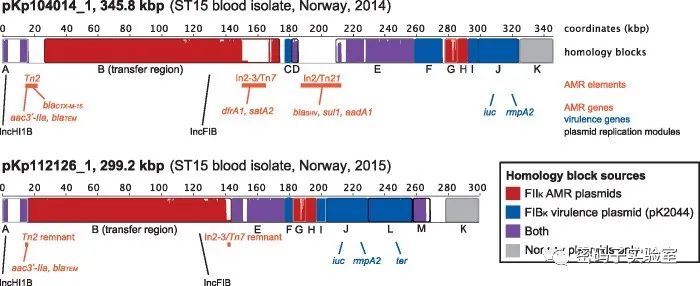
1. hv-MDR质粒图谱,显示了与使用Mauve生成的AMR(pKp_Goe_579-1)和毒力(pK2044)质粒密切相关的同源区域。
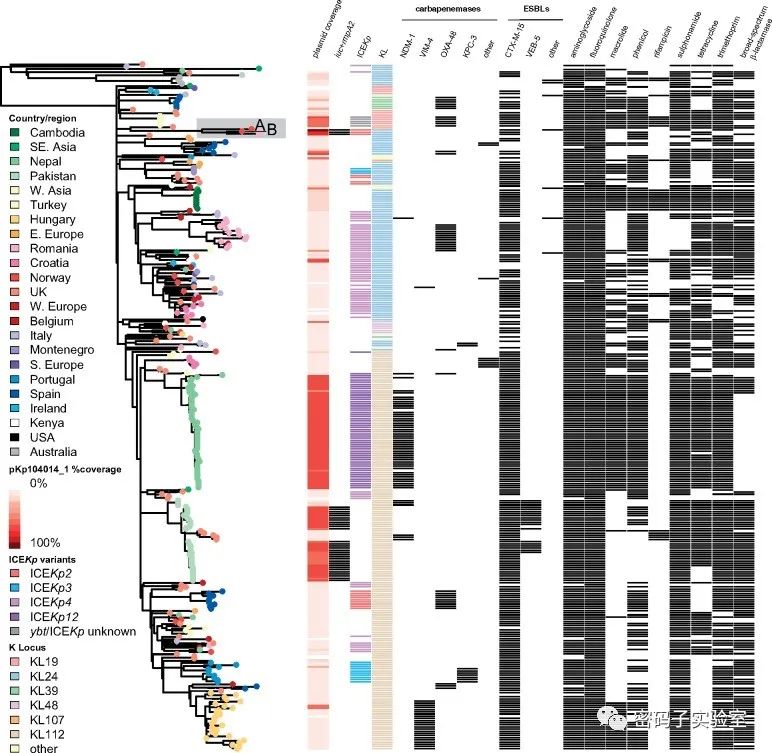
2.使用核心基因组SNP进化树分析,挪威镶嵌hv-MDR菌株KP_NORM_BLD_2014_104014和KP_NORM_BLD_2015_112126分别标记为A和B,并在灰色框中与相关的罗马尼亚分离株一起突出显示。显示这两个挪威hv-MDR分离株彼此密切相关(77个SNP,0.001%核苷酸差异),并与2013年从罗马尼亚采集的尿液分离物(110个SNP)密切相关,但相距甚远(差异>0.003%)来自其他挪威和全球隔离株。
>>
上海唯那生物专注于提供微生物包括耐药性研究在内的各类个性化服务,包括基金方案申报、组学测序、个性化生信分析、文章辅导发表、科研绘图、微生物纯化培养、基因克隆、微生物突变体(库)构建等。全方位,一站式,我们期待与您的合作。从数据处理、相关运算,到结果解读、图片绘制,全方位、一站式提供相关研究的解决方案,大家有需要的话也请联系我们。
>>
唯那生物精心准备的微生物耐药专题解析合集材料,具体包括:
1、微生物耐药研究快速入门综述;
2、微生物耐药研究经典案例解读;
3、微生物耐药研究专题方案书;
4、微生物耐药研究的热点好文章。

-
点赞 (0人)
- 收藏 (0人)



