细菌质粒复制子预测与结果解析
- 看不见的线
- 735
- 2025-04-30 10:17:36
- 原创
可用分析工具:
PlasmidFinder(https://cge.food.dtu.dk/services/PlasmidFinder/):依据质粒不相容性Rep typing(Inc)预测肠杆菌及部分革兰氏阳性菌的复制子(replicon)。
pMLST(https://cge.food.dtu.dk/services/pMLST/):预测IncF的亚型IncFIA、IncFIB、IncFIC、IncFII、IncFIIK、IncFIIS、IncFIIY及其亚等位基因编号。分析IncA/C、IncHI1、IncHI2、IncI1、IncN、pBSSB1-famil、Shigella flexneri复制子的序列分型,原理类似MLST。
KpVR(https://bioinfo-mml.sjtu.edu.cn/KpVR/index.php):在PlasmidFinder的基础上添加了IncFIB(K)的分型数据,适用于针对IncFIB(K) 的进一步研究。
其它综合性分析工具,如ABRicate、BacWGSTdb等大多整合了PlasmidFinder数据库的数据,可以作为PlasmidFinder的替代工具。
注意事项:
一个质粒可能含有多个复制子,属于正常现象;
PlasmidFinder数据库中部分早期复制子命名可能不够规范,在实际使用时,建议参考最新研究调整。
示例结果解读:
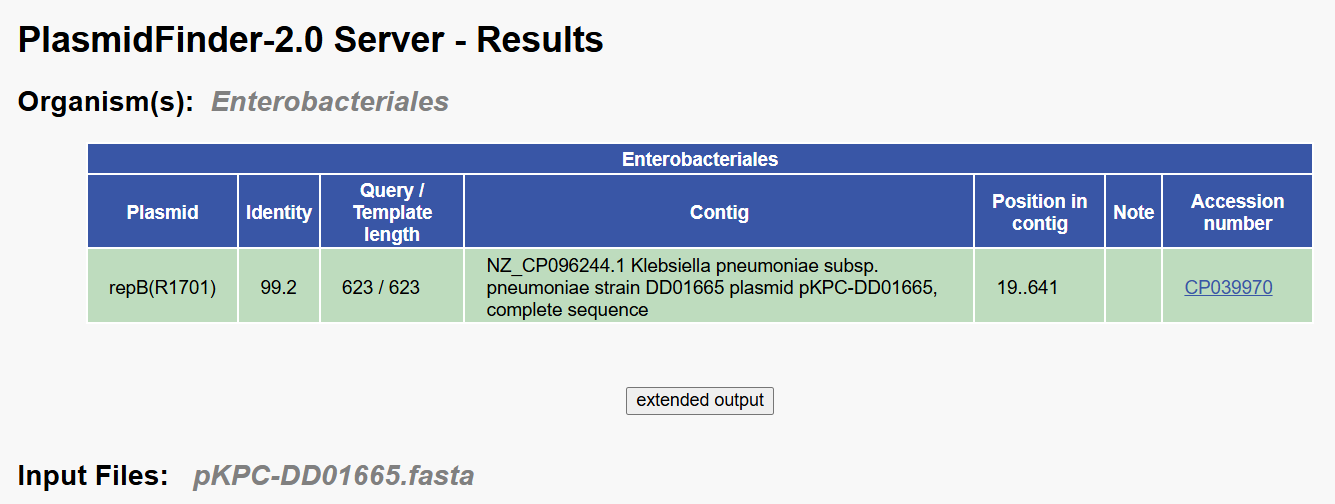
如下图所示,pKPC-DD01665是肺炎克雷伯菌DD01665所携带的质粒,PlasmidFinder预测显示该质粒仅含有一个复制子,类型为repB(R1701),identity为99.2%。
由于repB编码蛋白全称是RepB family plasmid replication initiator protein,原则上来说属于基因名称,不算复制子类型(Inc),因此最好替换为Inc命名法中的统一名称。
按照pMLST预测的结果,pKPC-DD01665复制子类型属于IncFIIK家族,与第34号等位基因IncFIIK34一致,这为复制子提供了更加合适的命名方法。

也有一些复制子采用两者的命名均可,如pHN7A8,它在PlasmidFinder中的复制子类型为IncFII(pHN7A8),因为它是这一类质粒中最早完成测序的提交样本;而在pMLST中,则为其单独分配了亚等位基因编号33,取名为IncFII33复制子,简写为IncF33。两者均可以表示IncFII复制子的情况。不过对于携带IncF型复制子的质粒,由于它们在各种病原菌中广泛存在,种类多样,结构组成复杂,且通常与耐药、毒力的传播有关,还是推荐大家使用pMLST做进一步亚型分析,不仅能提供更精细的等位基因分型信息,也有助于精准溯源和功能研究。
局限性说明:
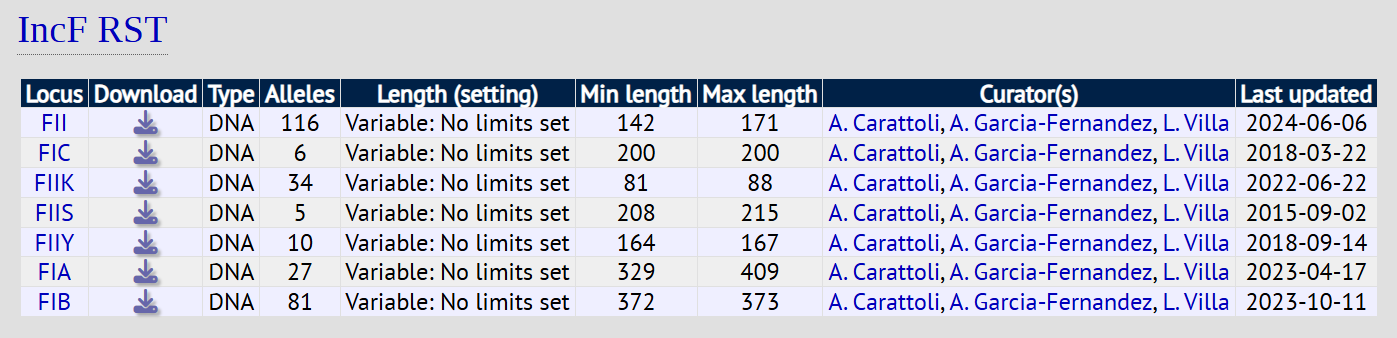
需要注意的是pMLST数据库收录的IncF等位基因数据仍然较为有限,某些IncF复制子可能无法预测到。在pubMLST数据库(https://pubmlst.org/)的Plasmid MLST条目中我们可以明确发现这一点(IncF RST)。
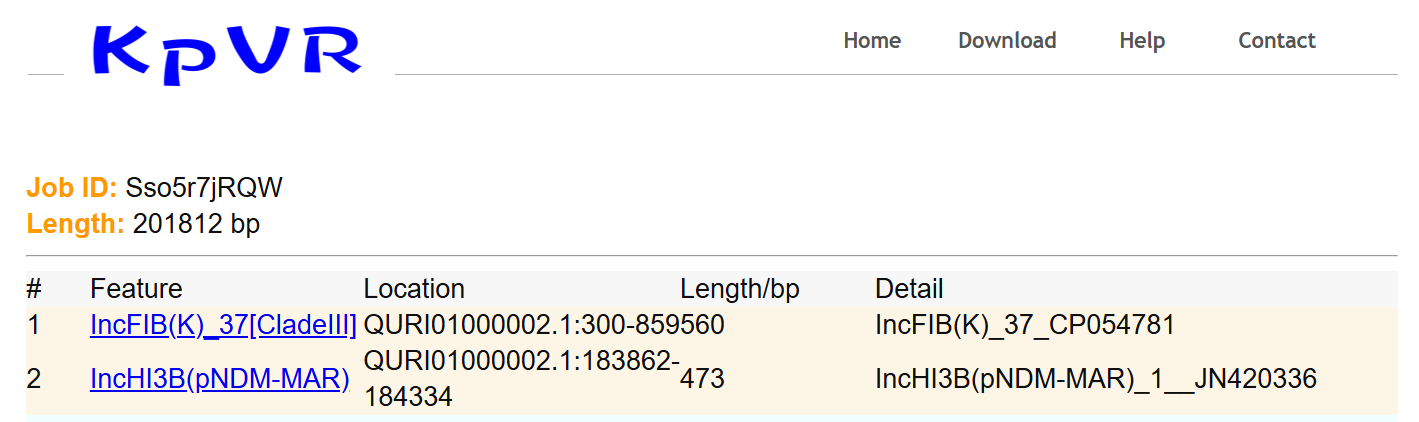
为此,上海交通大学开发的KpVR工具便针对IncFIB(K)复制子进行了扩展,其原理是利用 “IncFIBK_1-JN233704”作为参考,通过ClustalW多序列比对,赋予每一种SNP/Indel新的等位基因编号。如下图所示,PlasmidFinder显示pvir-KP47434_2的复制子包括repB和IncHI3B(pNDM-MAR),而在KpVR中,repB被明确为IncFIB(K)_37。
建议和总结:
1、 质粒复制子基础研究仅使用PlasmidFinder预测即可。
2、 若预测结果出现repA、repB等不明确的复制子类型或需要做较为细致的研究(主要针对IncF复制子),推荐联合使用pMLST进行分析。同时新等位基因,可以将其提交至pMLST数据库。
3、 数据库中未分型的质粒复制子,可以参考上述KpVR的方法,创建一种新的亚型命名。
文献推荐:
以下文献在质粒复制子分型研究时,使用了上述策略,可以参考阅读。
Tian D, Wang M, Zhou Y, Hu D, Ou HY, Jiang X. Genetic diversity and evolution of the virulence plasmids encoding aerobactin and salmochelin in Klebsiella pneumoniae. Virulence. 2021;12(1):1323-1333.
Jiang J, Wang L, Hu Y, et al. Global emergence of Carbapenem-resistant Hypervirulent Klebsiella pneumoniae driven by an IncFIIK34 KPC-2 plasmid. EBioMedicine. 2025;113:105627.
Sun Z, Zhang J, Wang C, et al. The pivotal role of IncFIB(Mar) plasmid in the emergence and spread of hypervirulent carbapenem-resistant Klebsiella pneumoniae. Sci Adv. 2025;11(5):eado9097.
-
点赞 (0人)
- 收藏 (0人)



