噬菌体的分类与命名:从传统形态到基因组学
- 看不见的线
- 43
- 2025-09-10 09:49:11
- 原创
第一部分:病毒分类与命名的通用规则
1、病毒分类的负责机构
病毒的分类和命名一般由国际病毒分类委员会(ICTV)负责,但ICTV不负责种以下的分类如血清型、基因型等,这些需要由国际专家小组确定。
2、病毒分类层级与最新统计
ICTV于2019年5月发布的病毒分类表将分类体系从“目”进一步扩展至“圈”,形成了目前共15个层级的分类系统。
每年ICTV都会多次对分类标准进行更新,最新版分类表“MSL40.v2”已于2025年8月发布,下表中列出了当前各分类层级的统计数量,详细内容可通过ICTV官方网站的 Master Species Lists (MSL)界面(https://ictv.global/msl)下载查看。当病毒可明确归入某科但属未确定时,称为该科的未确定种(unassigned species)。
| 中文名 | 英文名 | 病毒后缀 | 数量 |
| 圈 | Realm | -viria | 7 |
| 亚圈 | Subrealm | -vira | 0 |
| 界 | Kingdom | -virae | 11 |
| 亚界 | Subkingdom | -virites | 0 |
| 门 | Phylum | -viricota | 22 |
| 亚门 | Subphylum | -viricotina | 4 |
| 纲 | Class | -viricetes | 49 |
| 亚纲 | Subclass | -viricetidae | 0 |
| 目 | Order | -virales | 93 |
| 亚目 | Suborder | -virineae | 12 |
| 科 | Family | -viridae | 368 |
| 亚科 | Subfamily | -virinae | 213 |
| 属 | Genus | -virus | 3768 |
| 亚属 | Subgenus | -virus | 86 |
| 种 | Species | 16213 |
此外,ICTV还为每个病毒物种指定了一个或多个“范例病毒”作为标准参考,相关数据可在Virus Metadata Resource (VMR)数据库(https://ictv.global/vmr)中获取。
第二部分:噬菌体的分类系统与命名方法
1、传统分类方法
传统分类主要依据噬菌体的核酸类型和形态结构(通过电镜观察)。
研究表明,超过95%的已分离噬菌体具有线性双链DNA(dsDNA)基因组,并被包装于带尾的蛋白质衣壳中。其余噬菌体在结构上呈现多样性:一部分虽含dsDNA基因组但缺乏尾部结构,另一部分则携带单链DNA(ssDNA)或RNA基因组,且同样不具有尾部。
在早期分类中,David Bradley根据尾部形态将有尾噬菌体划分为三类:
①肌尾,对应肌尾噬菌体科 Myoviridae,以T4 phage为代表;
②短非收缩尾,对应短尾噬菌体科 Podoviridae,以T7 phage为代表;
③长非收缩尾,对应长尾噬菌体科 Siphoviridae,以λ phage为代表。
目前这三类已被统一归类于有尾病毒纲(Caudoviricetes)。
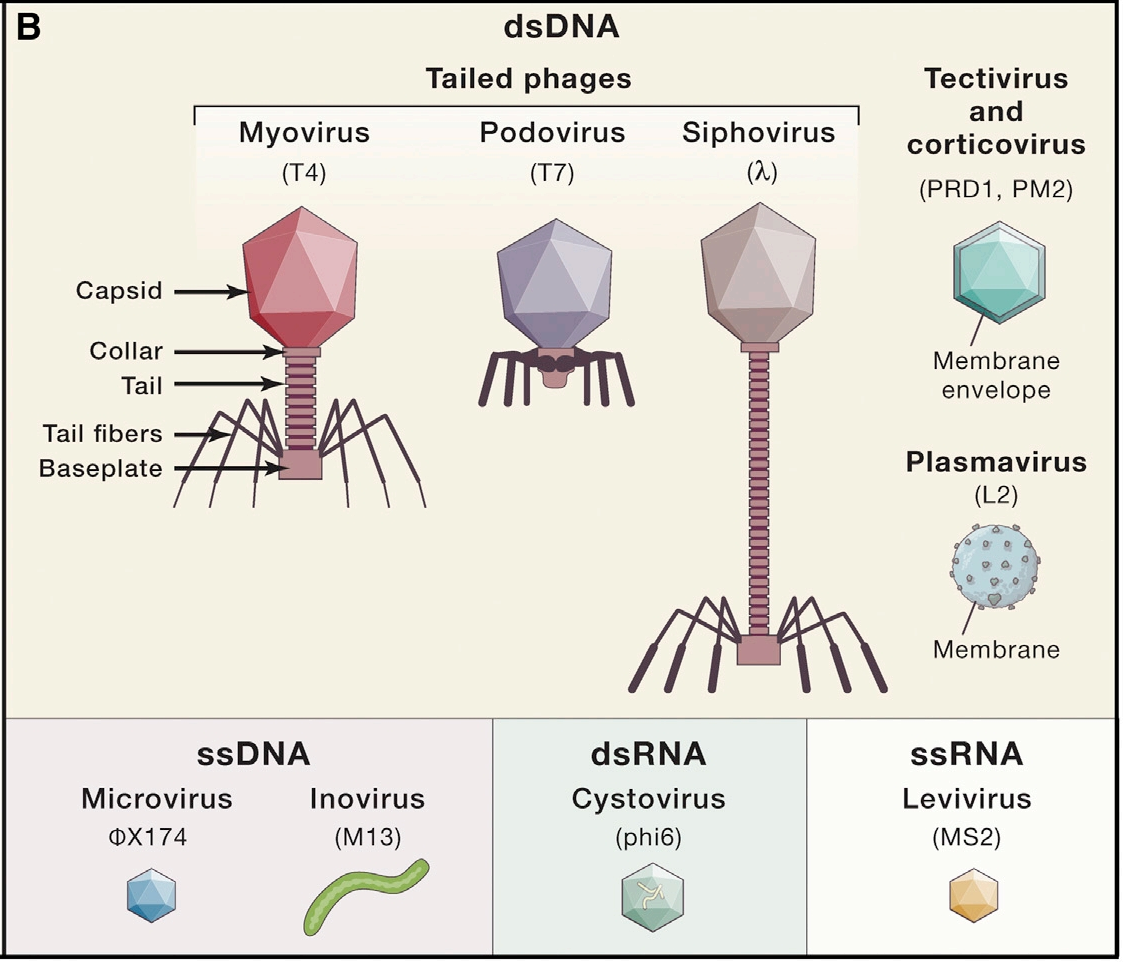
图1噬菌体传统分类
2、系统发育更新
随着比较基因组学和噬菌体全基因组测序技术的迅猛发展,利用系统发育分析对噬菌体进行分类已成为一种更为精准的策略。该方法不仅可基于全基因组序列,也常借助于保守蛋白序列构建进化树,从而更细致地解析噬菌体间的进化关系。它有效补充并改变了以往依赖形态学、宿主范围等有限特征的传统分类与命名体系,推动噬菌体分类系统向着更科学、更系统化的方向更新与发展。
如下文所示,为一种新型多价噬菌体S144。作者首先通过BLASTn比对将其全基因组与GenBank数据库进行比对,初步结果显示S144与沙门氏菌噬菌体ZCSE2和SE4高度相似(后两者是Loughboroughvirus属的成员),与新建立的Rosemountvirus属亲缘关系较近。
在此基础上,为更准确判定S144的分类地位,作者进一步构建了一个包含97个噬菌体基因组的数据库,其中包括S144及其近缘物种、感染肠杆菌科的各属代表株,并以人类疱疹病毒作为外群。利用VICTOR在线分析平台(https://victor.dsmz.de),基于全基因组或蛋白质组数据构建系统发育树,结果明确S144归属于Loughboroughvirus属。(ViPTree(https://www.genome.jp/viptree/)是另一常用病毒进化树构建工具,可基于tBLASTx计算全基因组序列相似性生成蛋白质组树)
为进一步验证这一分类结果,作者还额外比对了数据库中已注释噬菌体的四种高度保守蛋白(包括主要衣壳蛋白、DNA聚合酶、门户蛋白和末端酶大亚基),并据此构建了系统发育树,该分析同样支持将S144划归Loughboroughvirus属。
值得注意的是,由于Loughboroughvirus属与Rosemountvirus属之间显示出高度基因组相似性、明显的共线性(基于Easyfig)以及保守蛋白序列一致性,作者进一步提出将这两个属共同归为一个新的亚科,命名为“Salusvirinae”。
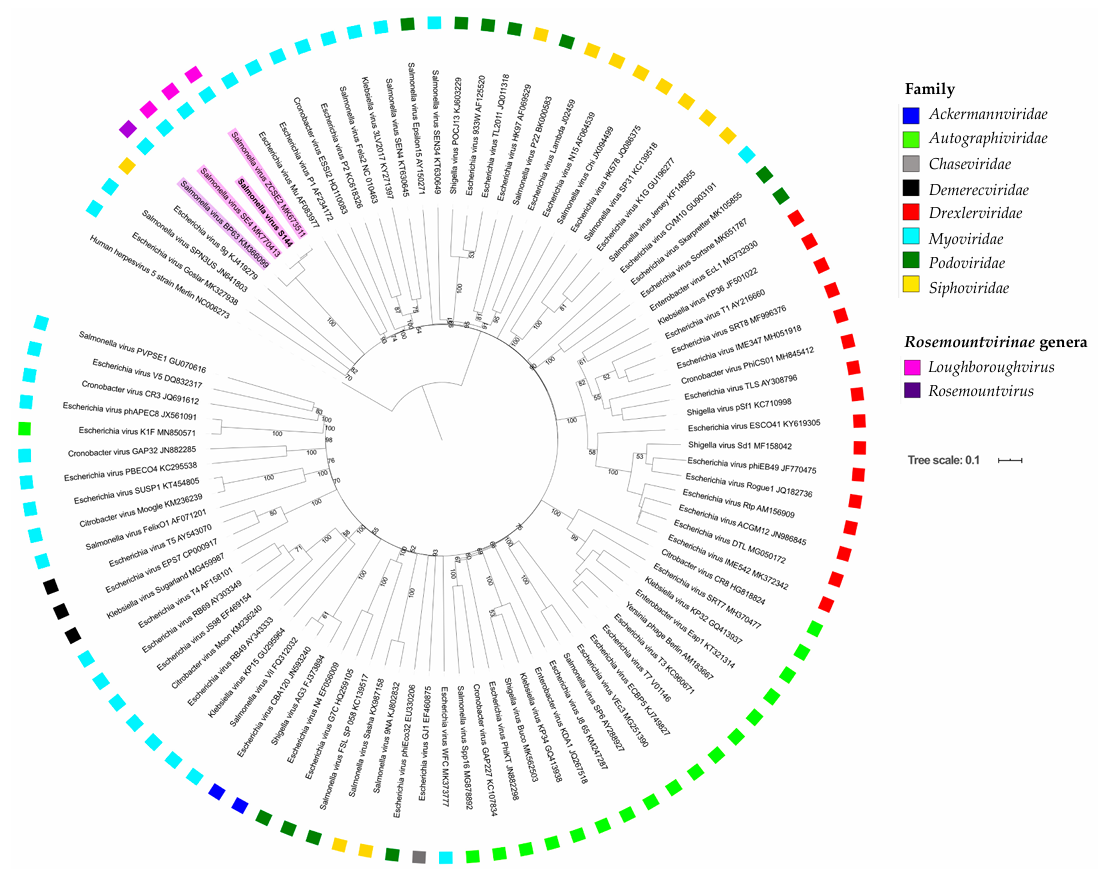
图2 VICTOR-噬菌体全基因组核苷酸水平的系统发育分析

图3 CLC Workbench-噬菌体a.主要衣壳蛋白、b. DNA聚合酶、c.门户蛋白和d.末端酶大亚基的蛋白质系统发育树
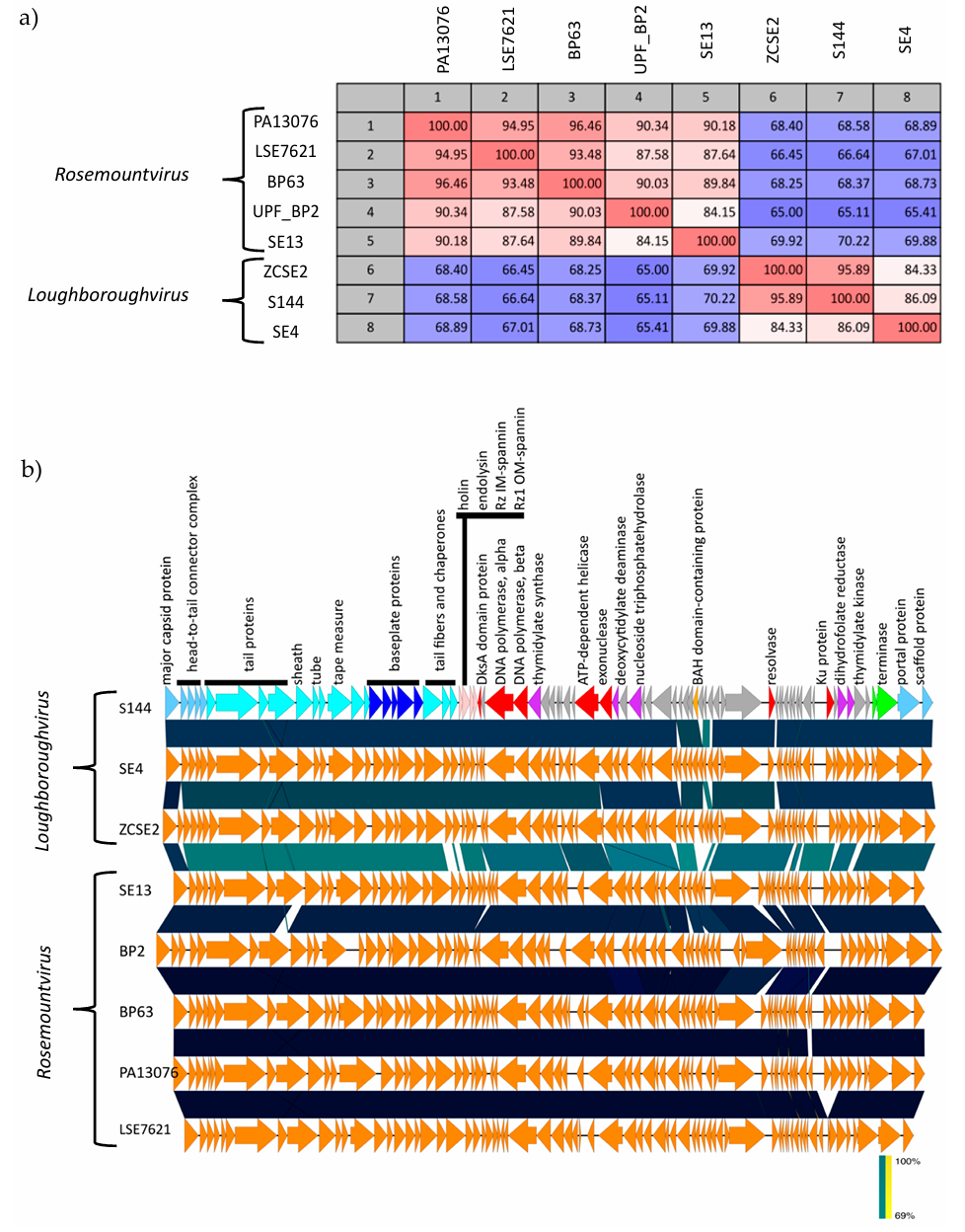
图4 a.CLC Workbench-核苷酸序列同一性矩阵;b. Easyfig-噬菌体全基因组线性比较
第三部分:新噬菌体的鉴定、分类与命名流程
1、噬菌体命名格式
一般由3部分组成:宿主名 + virus/phage +独特名(e.g.,Escherichia virus T4)。
更多内容请参考文档-The International Code of Virus Classification and Nomenclature (ICVCN; the ICTV Code)(https://ictv.global/about/code)
2、如何确定新噬菌体属于哪个科、属或种?
早期分类在一定程度上可能依赖于经验性的阈值,例如Adriaenssens E(2017)在文献中提出的建议:
- 科(Family)水平:主要依据形态特征(如电镜下的尾部结构)。
- 属(Genus)水平:与已知属的成员基因组序列相似性大于50%,可归为同一属。
- 种(Species)水平:与已知噬菌体的基因组序列差异小于5%,可归为同一种。
但这并非ICTV的正式规定,一般只作为初步筛选的参考,特别是对于属的确定,50%的阈值可能并不够准确。例如,病毒基因组间距离计算工具VIRIDIC(https://rhea.icbm.uni-oldenburg.de/viridic/)便默认以95%和70%分别作为种和属的建议性阈值。
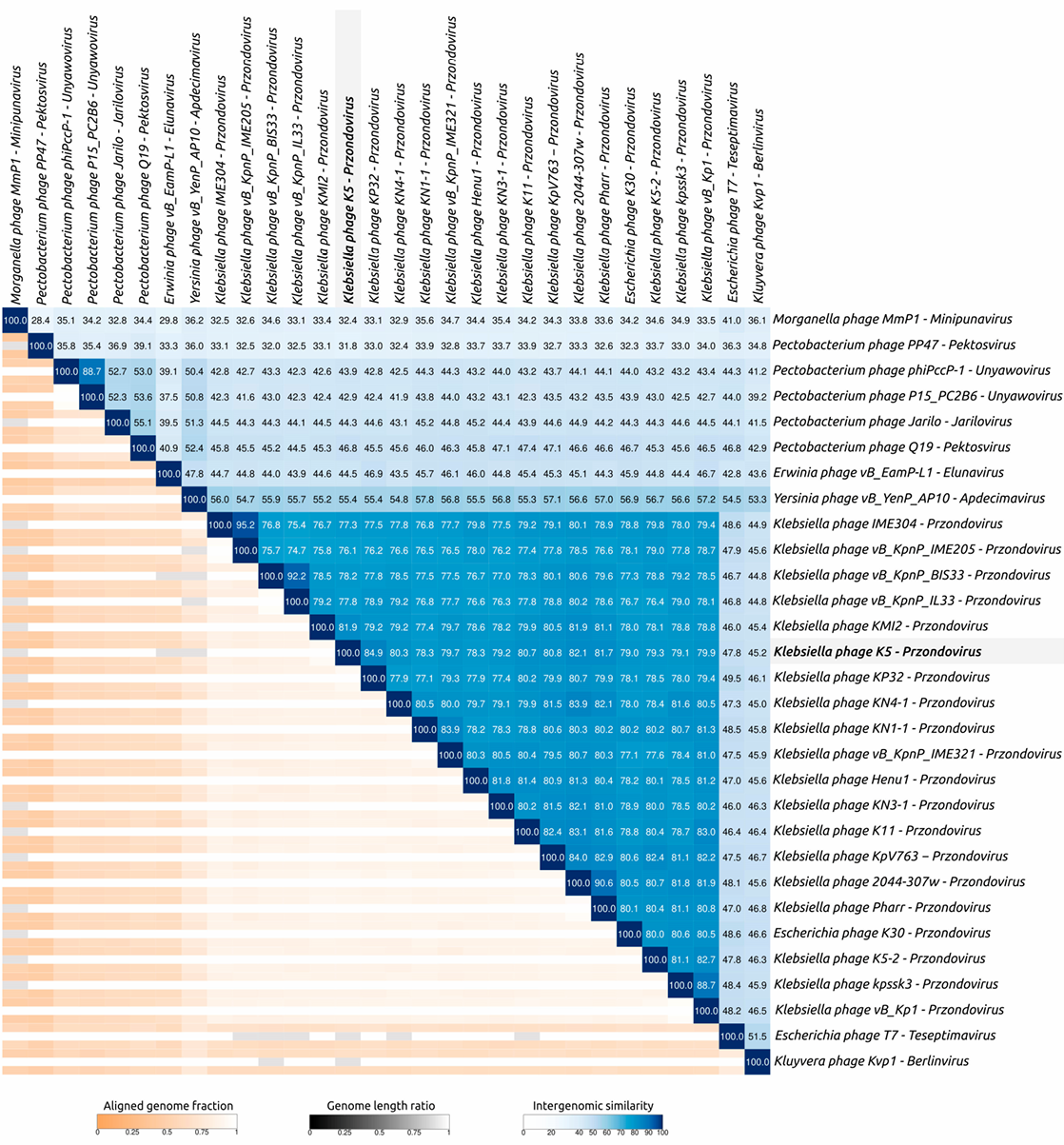
图5 VIRIDIC-基因组核苷酸相似性热图
ICTV建议采用多方面的证据而非依靠单个指标来决定一个病毒(包括噬菌体)的分类地位,通常可以考虑综合以下三方面的证据:
①系统发育分析(核心依据):基于全基因组序列或一组保守的标记基因(如主要衣壳蛋白、终止酶大亚基)构建稳健的系统发育树。属(Genus)水平的确定尤其依赖于新噬菌体能否与已知属的成员形成一个稳定的“单系群”(即拥有一个共同的祖先且所有后代都包含在内)。
②基因组序列相似性(重要参考):平均核苷酸同一性(ANI)、平均氨基酸同一性(AAI)等数值仍是重要的量化参考,主要用于支持系统发育分析的结果,并在种(Species)水平界定中权重较高。
③病毒的基本特征(辅助证据):包括基因组特性、病毒粒子结构、基因表达与复制策略以及宿主范围与致病性等。这些特征有助于确认分类地位并揭示其生物学特性。
参考文献
[1]胡福泉,童贻刚.噬菌体学:从理论到实践[M].北京:科学出版社, 2021: 51-53.
[2] Adriaenssens E, Brister JR. How to Name and Classify Your Phage: An Informal Guide. Viruses. 2017;9(4):70. doi:10.3390/v9040070
[3] Ofir G, Sorek R. Contemporary Phage Biology: From Classic Models to New Insights.Cell. 2018;172(6):1260-1270. doi:10.1016/j.cell.2017.10.045
[4] Gambino M, Nørgaard Sørensen A, Ahern S, et al. Phage S144, A New Polyvalent Phage Infecting Salmonella spp. and Cronobacter sakazakii. Int J Mol Sci. 2020;21(15):5196.doi:10.3390/ijms21155196
[5] Meier-Kolthoff JP, Göker M. VICTOR: genome-based phylogeny and classification of prokaryotic viruses. Bioinformatics. 2017;33(21):3396-3404. doi:10.1093/bioinformatics/btx440
[6] Nishimura Y, Yoshida T, Kuronishi M, et al. ViPTree: the viral proteomic tree server. Bioinformatics. 2017;33(15):2379-2380. doi:10.1093/bioinformatics/btx157
[7] Moraru C, Varsani A, Kropinski AM. VIRIDIC-A Novel Tool to Calculate the Intergenomic Similarities of Prokaryote-Infecting Viruses. Viruses. 2020;12(11):1268. doi:10.3390/v12111268
[8] Depolymerisation of the Klebsiella pneumoniae Capsular Polysaccharide K21 by Klebsiella Phage K5. Int J Mol Sci. 2023;24(24):17288. doi:10.3390/ijms242417288
-
点赞 (0人)
- 收藏 (0人)
-
上一篇: 没有了
- 下一篇: 微生物基因组研究:新种鉴定、进化分型和比较分析



