跟着Nature学细菌耐药基因分析:实时基因组学检测隐藏的抗生素耐药性
- 看不见的线
- 1545
- 2025-02-11 11:50:58
- 文章来源:简说基因
克雷伯菌耐药性严重,尤其是耐碳青霉烯类菌株广泛传播,导致常规治疗失效和患者死亡风险增加。研究其耐药机制和开发新型药物迫在眉睫,以应对这一公共卫生威胁。今天我们就一起来学习下面这篇关于克雷伯菌耐药性文章的研究思路和生信分析方法。

这篇文章的生信分析部分用到了Minimap2、SAMtools等工具,这些工具都能在Galaxy生信云平台(网址:usegalaxy.cn )上方便地使用,不需要安装软件和配置复杂的环境,也无需下载大型的耐药基因数据库。有兴趣的读者可以参考以下文章:
一文解锁Samtools一文详解细菌耐药性生信分析:从下机数据到耐药基因鉴定
下面,我们就跟随Nature的步伐,一起深入探索细菌耐药性的奥秘。
摘要:纳米孔测序的实时基因组学有望在临床环境中快速预测抗生素耐药性。本文通过肺炎克雷伯菌感染病例,展示了该技术如何提高复杂感染中抗生素耐药性分析的准确性,能检测到传统方法未发现的低丰度质粒介导的耐药性,对临床决策和患者预后具有重要意义。
一、引言
世界卫生组织已将抗生素耐药性列为十大全球最严重的健康威胁之一,耐药感染导致死亡率和发病率升高,因为延迟或不适当的治疗。快速准确识别耐药细菌病原体可以促进早期适当治疗,降低死亡率和感染及治疗相关的发病率。纳米孔测序技术推动的实时基因组学有望在临床环境中加快病原体识别和抗生素耐药性分析。然而,其预测抗生素耐药性的准确性仍需与传统诊断方法直接比较,以纳入常规临床实践。本文通过肺炎克雷伯菌感染病例,展示实时基因组学在临床中的优势。
二、结果
研究设计
- •对比分析:使用同一感染病例的细菌分离株,对临床诊断方法和实时基因组学预测进行对比分析(图1)。
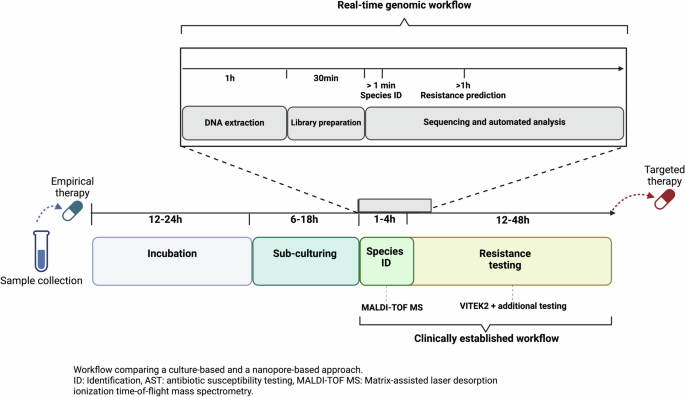
- 图1:实时基因组学(顶部)和临床常规(底部)用于病原体物种识别和抗生素耐药性分析的工作流程概述
- •病例介绍:德国一免疫功能低下的患者出现发热,初始用美罗培南治疗,后根据临床诊断更换为头孢他啶 - 阿维巴坦,但患者病情恶化。
临床诊断结果
- •初始诊断:气管吸取物样本检测出耐碳青霉烯的肺炎克雷伯菌,推测其耐药机制为肺炎克雷伯菌碳青霉烯酶(KPC)。
- •治疗调整:诊断显示头孢他啶 - 阿维巴坦敏感,治疗改为该药物,但患者病情恶化。
- •后续诊断:血液培养样本显示肺炎克雷伯菌对碳青霉烯敏感性恢复,但对头孢他啶 - 阿维巴坦耐药,临床诊断未检测到新的碳青霉烯酶。
实时基因组学诊断结果
- •测序与分析:使用便携式纳米孔测序设备Mk1b对治疗前后的肺炎克雷伯菌分离株进行快速测序和分析,包括DNA提取、文库制备、测序和自动分析。
- •病原体鉴定与耐药预测
病原体鉴定:正确鉴定出治疗前后的菌株均为肺炎克雷伯菌。
耐药基因检测
1. 前处理菌株:检测到准确(>90%)的blaKPC - 2基因拷贝,证实了临床诊断的美罗培南耐药性。
2. 后处理菌株:检测到大量的blaKPC - 14基因拷贝,该基因此前被认为是少数能导致头孢他啶 - 阿维巴坦耐药同时可能恢复对碳青霉烯敏感性的KPC亚型,基因组学预测确定了blaKPC - 14为潜在的耐药机制。
3. 早期检测潜力:实时基因组学方法还检测到前处理菌株中存在单拷贝的blaKPC - 14抗性基因,额外测序显示在短时间内可检测到更多基因拷贝,快速提示头孢他啶 - 阿维巴坦耐药的可能性(图2)。
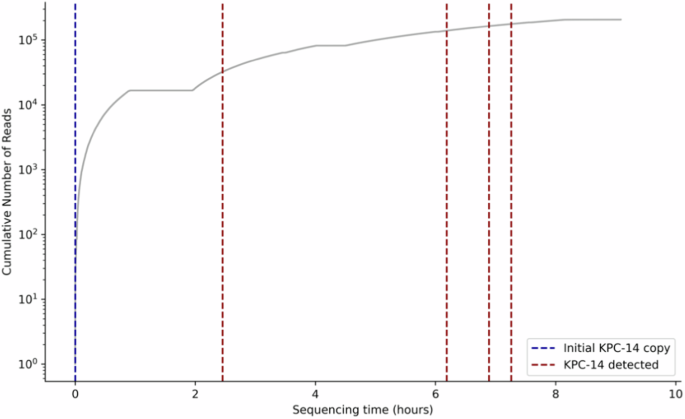
图2:在临床环境下对治疗前细菌分离株的技术重复样本进行适应性测序运行模拟期间,blaKPC - 14拷贝数检测的时间线。
- •流行病学和功能分析
基因组组装:成功组装出治疗前后菌株的染色体和质粒基因组(图3)。
序列类型分析:核心基因组多位点序列分型(cgMLST)显示两个菌株均为新兴的高危序列型ST147,且与全球已知的ST147基因组遗传不同。
质粒分析
1. 质粒特征:blaKPC - 2和blaKPC - 14基因位于IncN质粒上,这些质粒高度同源,具有关键质粒特征,如松弛酶类型、转移起始位点和配对形成基因等,且具有接合性。
2. 质粒拷贝数:推断治疗前后菌株中IncN质粒相对于细菌染色体的拷贝数分别为3和4。
3. 耐药基因丰度变化:CAZ - AVI暴露后,blaKPC - 14基因的标准化丰度从0.56%增加到26.6%。
新KPC亚型鉴定:将后处理菌株提交给德国国家革兰氏阴性菌参考中心,最初定义的blaKPC - 14基因被鉴定为新的KPC亚型blaKPC - 159。

图3:A. 治疗前和B. 治疗后肺炎克雷伯菌分离株的基因组和质粒从头组装
三、讨论
技术优势
- •感染分析潜力:实时基因组学在该患者病例中的应用表明,该技术有望快速准确地分析复杂细菌感染,揭示临床诊断未发现的头孢他啶 - 阿维巴坦耐药性,影响治疗决策。
- •公共卫生意义:基因组数据能追踪患者感染中质粒拷贝数变化与抗生素耐药性的关系,多个质粒的存在可能加速耐药性出现,对有效治疗策略构成挑战。
研究局限性
- •样本局限性:仅关注细菌分离株可能无法完全了解患者感染的微生物多样性,使用直接患者样本的宏基因组测序可能会发现更多影响抗生素耐药性的因素。
- •样本量局限性:研究仅涉及同一患者的两个细菌分离株,无法深入了解质粒选择如何应对抗生素选择压力的变化。
四、方法
临床诊断流程
- •样本处理:临床样本在BD® Columbia Blood和MacConkey琼脂平板上培养,然后进行亚培养,使用MALDI - TOF MS进行物种鉴定。
- •抗生素敏感性测试:使用VITEK2进行抗生素敏感性测试,对于碳青霉烯耐药的菌株,使用多重免疫色谱测定法检测碳青霉烯酶,使用Liofilchem® MIC测试条测量头孢他啶 - 阿维巴坦的MIC。
实时基因组学数据生成
- •样本处理:储存的菌株解冻后在BD® Columbia血液琼脂平板上培养,提取DNA。
- •测序步骤
第一轮测序:使用Maxwell® RSC Blood DNA提取试剂盒提取DNA,使用SQK - RBK004快速条形码试剂盒制备纳米孔测序文库,在Oxford Nanopore Technologies MinION MK1b设备上测序15小时。
第二轮测序:对前处理菌株进行二次培养,提取DNA并测序8小时。
实时基因组学数据分析
- •数据处理软件:使用Guppy进行碱基调用,Porechop修剪适配器序列,Nanofilt过滤低质量和短序列,NanoStat生成测序总结。
- •分析流程
基本分析:使用EPI2ME的Antimicrobial Resistance蛋白同源模型进行质量控制、物种鉴定和抗生素耐药性预测。
基因组组装:使用Flye进行从头组装,使用Minimap2和Racon进行抛光,使用SAMtools depth评估组装覆盖度,使用Pathogenwatch进行物种复杂分析和核心基因组多位点序列分型。 - •质粒检测与注释
可视化与鉴定:使用Bandage可视化组装图,识别染色体和质粒基因组,使用MOB - suite和mobileOG - db进行质粒功能注释。
拷贝数估计:通过计算质粒复制子测序深度与染色体重叠群测序深度的比值来估计质粒拷贝数,通过提取特定读段ID和比较抗性基因与最丰富抗性基因的拷贝数比值来准确估计抗性基因的标准化丰度。 - •参考中心注释:将后处理细菌分离株提交给德国国家革兰氏阴性菌参考中心,鉴定出blaKPC - 14基因变体为新的KPC亚型blaKPC - 159。
-
点赞 (0人)
- 收藏 (0人)



