最小生成树构建软件Grapetree与Phyloviz的差异
- 看不见的线
- 2247
- 2023-12-14 08:54:36
- 原创
Grapetree包含四种算法:MSTreeV2、MSTree、NJ、RapidRJ。
根据开发者测试的结果,NJ/RapidNJ拥有最高的特异性(specificity)和灵敏度(sensitivity);goeBURST、dMST的特异性受等位基因距离和丢失数据的比例影响较大,其灵敏度略高于MSTree V2;MSTree V2始终保持高特异性和低灵敏度,但是对数据缺失样本的分析结果相对较好。(在此结果中,低特异性代表假阳性,低灵敏度代表假阴性偏高)
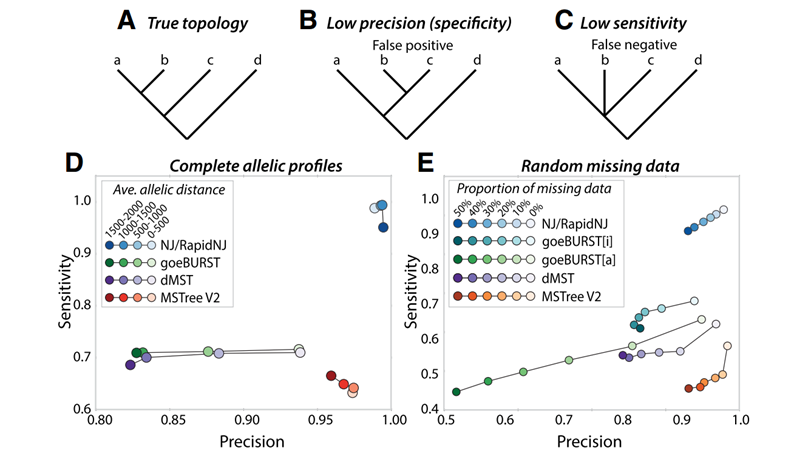
在最小生成树(minimum spanning tree, MST)构建过程中,Bionumics或goeBURST依据eBURST原则通过tie-breaking在多个共优分支之间进行选择。eBURST方法假定克隆复合体(clonal complex, CC)是由founder基因型建立的,该founder基因型的遗传变异反映了随着时间的推移额外变异的逐步积累。另一隐藏的含义是变异的数量随着与founder基因型距离而减少,这样founder基因型就等同于具有最多单位点变异的中心基因型。
MSTree V2不依赖于理论founder基因型的早期属性,而是简单地根据等位基因距离的调和平均值在多个共优分支之间选择中心节点。在该算法中,给定了一个centroid genotype的概念,即对于任意给定群体中的基因型与该群体中的其它基因型必定有最小的等位基因距离。等位基因的调和平均值公式为:
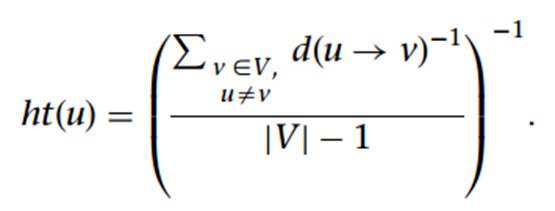
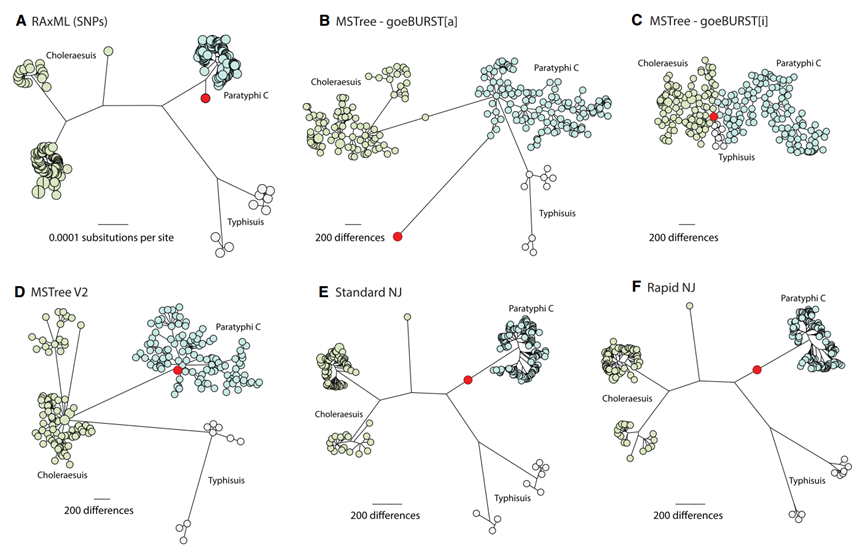
所以Grapetree和Phyloviz由于计算方法的不同,在绘制最小生成树时,两者的结果可能存在一定的差异,这都是正常的情况。
参考文献:
Zhou Z, Alikhan N-F, Sergeant M J, et al. GrapeTree: visualization of core genomic relationships among 100,000 bacterial pathogens [J]. Genome Research, 2018, 28(9): 1395-404.doi: 10.1101/gr.232397.117
推荐课程
【课程】微生物比较基因组精品系列课——全套自学必入的系统课程
课程链接:微生物比较基因组精品系列课【全套】
【课程】微生物比较基因组与群体进化——基因组变异专题研究
课程链接:微生物比较基因组与群体进化
【课程】微生物分子分型-MLST课程——分型全套(含理论、软件、方法)
课程链接:微生物分子分型-MLST课程
【课程】基因组结构分析神器Easyfig实操精品课
【课程】BRIG绘图——结构比较专题2
【课程】肺炎克雷伯菌基因组学研究综合指南
课程链接:肺炎克雷伯菌基因组学研究综合指南
【课程】微生物基因组生信必学课程
课程链接:微生物基因组生信分析必学课程
【课程】微生物生防菌研究
课程链接:生防菌的系统化研究
专题材料
【资料】耐药专题材料
【资料】生防专题材料
请添加唯那生物技术客服小唯的微信号winnerbio01,备注“耐药专题”或“生防专题”,立马获取。
更多专题推荐
CORPORATE CULTURE
1、耐药毒力专题
-
点赞 (0人)
- 收藏 (0人)



