囊性纤维化患者气道中铜绿假单胞菌谱系基因的丢失和获得
- 看不见的线
- 787
- 2024-01-24 10:19:36
- 原创
文章题目:Gene Loss and Acquisition in Lineages of Pseudomonas aeruginosa Evolving in Cystic Fibrosis Patient Airways
文章链接:doi: 10.1128/mBio.02359-20
发表时间:2020.10
发表期刊:mBio
IF:6.4
研究背景:基因的获得和丢失对于细菌的进化和新环境适应非常重要。与点突变、微插入缺失、倒置易位等逐渐改变现有基因组成分的方式相比,整个基因的获得或丢失会产生更加迅速的变化,并直接影响细菌的表型,如毒力、抗生素耐药性和代谢能力等等。为更好地了解铜绿假单胞菌(Pseudomonas aeruginosa)进化和适应性发展过程的遗传基础,本文作者对P. aeruginosa从最初入侵囊性纤维化(cystic fibrosis, CF)患者气道到适应人类宿主微环境的基因丢失和获得情况展开了研究。
研究对象:囊性纤维化患者铜绿假单胞菌分离株基因组序列(446株)
研究方法:全基因组测序、克隆型鉴定、泛基因组分析、移动元件分析、毒力因子分析、趋同进化分析、功能注释、单核苷酸多态性分析、系统发育分析。
主要研究内容及结果:
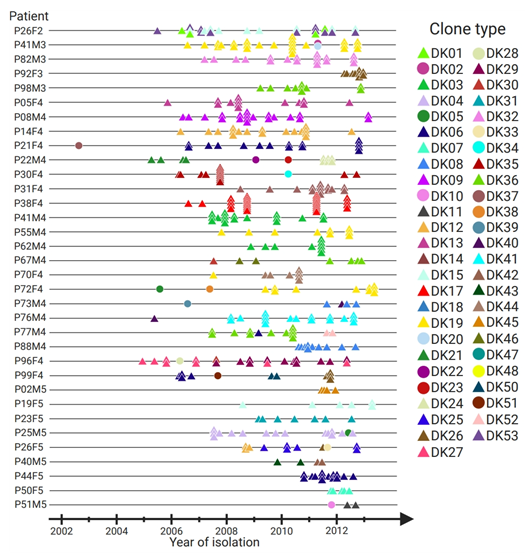
- 分离株基本信息及克隆型定义
本研究从34名CF患者的气道中采集了474株铜绿假单胞菌,并进行了基因组short-read测序,以探究患者P. aeruginosa感染后的0~9年内气道细菌谱系的基因组进化。通过基因组de novo组装,作者成功组装完成了446株菌(scaffold≤500),并以此作为样本基础鉴定在感染过程中丢失和获得的基因。
2015年该作者的一项研究中曾采用了一种ad hocanalysis的方法以定义不同P. aeruginosa分离株的克隆类型(依据SNP差异)。本研究进一步通过开发的Pactyper工具(https://github.com/MigleSur/Pactyper)来确定菌株的克隆类型(clone type)(两者原理类似)。Pactyper是在默认设置下运行的,如果两个分离株样本在整个群体所定义的全部核心基因组中SNP差异数目超过5000个SNPs,它们即被认为属于不同的clone type。且来自同一患者的相同克隆类型的分离株被认为属于同一谱系(lineage)。(对于lineage和clone type的关系可以参见图1)
最终该研究将446个分离株基因组分为了51个clone type,对应于先前研究中的DK01~DK53。并归为45个lineage,以便鉴定每位宿主体内P. aeruginosa积累的基因差异。总的来说,45个lineage包含423个分离株,分布在34名患者中,其中9名患者感染了两种或更多种clone type的P. aeruginosa,其余23个不包含lineage或仅有单个clone type的菌株被排除在分析之外。
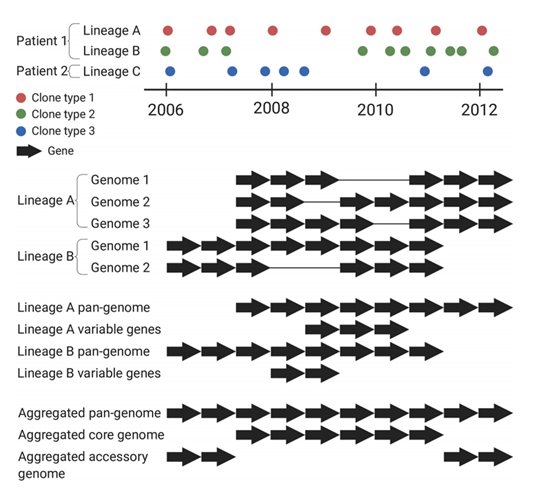
- 泛基因组和基因获得与丢失
作者分两步对423个基因组进行了泛基因组分析,以确定在谱系内或谱系间表现出变异的基因。
首先对同一谱系分离株的基因组进行了比较,以确定全套非冗余基因,即谱系泛基因组(lineage pan-genome)。谱系泛基因组包括两个部分:(i)存在于各自谱系所有分离株中的基因(lineage core genome);(ii)仅存在于部分谱系分离株中的基因(lineage variable genome),即在感染期间丢失或获得的基因。
其次,对谱系泛基因组进行了比较,以确定在所有谱系中发现的非冗余基因的完整集合,即聚合泛基因组(aggregated pan-genome)。聚合泛基因组包括由不同谱系泛基因组共有基因(aggregated core genome)和仅存在于某些谱系泛基因组中的基因(aggregated accessory genome)构成。结果显示大约一半的aggregated accessory genes(4932)是谱系所独有的。
此外,aggregated accessory genes在谱系内的变异频率是aggregated core genes的15倍。一个可能的解释是aggregated accessory genes携带大量的可移动遗传元件(MGE),如前噬菌体的origin序列。作者利用ACLAME数据库分别对aggregated core genes和aggregated accessory genes中的噬菌体和前噬菌体序列进行鉴定和注释,结果显示aggregated accessory genes含有116倍的前噬菌体基因,且这些基因高度可变。

- 感染过程中谱系基因含量的变化
接下来,为确定variable genes是来源于lineage的基因丢失还是基因获得,作者进一步对基因丢失(gene loss)和基因获得(gene acquisition)进行了定义。
loss,即在初始分离株中存在,在后续1个或多个分离株中不存在;acquisition,即在初始分离株中不存在,在后续1个或多个分离株中存在。(注意这种定义可能不够准确,因为初始分离株可能无法代表lineage最近的共同祖先)根据此定义,最终结果显示基因丢失的发生频率至少是基因获得的6倍。而在一个群体中基因丢失或获得的频率是单个基因的25倍,也就是说一个基因的丢失或获得通常伴随着其它基因的丢失或获得。
ACLAME数据库显示在45个lineage均有发现前噬菌体基因,其中22个lineage的可变基因中有前噬菌体基因,70%的病例中前噬菌体基因缺失。也就是说,前噬菌体在感染早期分离株中存在,而在后期分离株中不存在。相比之下,质粒基因在任何lineage中都未发现丢失或获得(基于PlasmidFinder数据库复制子定义质粒基因)。
VFDB数据库显示aggregated accessory genome共有257个基因与毒力相关,且其中17个基因至少在某个谱系中是可变的,8个基因在多个谱系中可变。two-sided Fisher's 精确检验表明,毒力基因丢失或获得的频率通常低于其他基因。Resfinder数据库,在aggregated accessory genome中确定了7个与抗生素耐药性相关的基因,且这些基因在任何谱系中均未发生丢失或获得。而在该作者此前报道的52个致病性适应基因中,有9个在lineage中发生了基因丢失或获得。two-sided Fisher's精确检验表明,病理适应性基因和非病理适应性基因的丢失或获得无显著差异。
- 趋同进化:相同基因在不同谱系中可变
虽然variable genes仅占aggregated accessory genome的一小部分,但研究者观察到某些基因在多个谱系中都是可变的。这里将在4个及以上的谱系中确定为可变的基因,定义为高度可变基因。在人工确认的52个variable genes中,存在于4个lineage的variable genes有47个,5个lineage有4个,10个lineage的仅有1个。
根据PseudoCAP功能分类和BLAST比对,作者对上述高度可变基因进行了功能注释。如图可见大多数高度可变的基因(34个)是假定功能基因或非假单胞菌起源的基因。第二大高可变基因组编码膜蛋白(4个)。其他高度可变基因编码蛋白质涉及氨基酸和核苷酸生物合成、抗生素耐药性和敏感性、转运、分泌因子、转录调节和代谢等等。

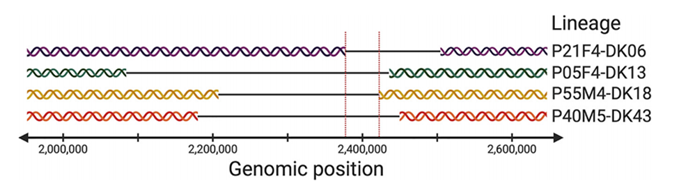
- hcnABC和exoY基因的趋同进化
在4个谱系(见图4)中,有34个基因出现了丢失或获得的情况,且这34个基因是PAO1参考基因组中PA2161 ~ PA2181基因和PA2189 ~ PA2204基因的同源基因。
34个基因中有17个被PseudoCAP注释为“假定的、未知的或未分类的”;其他基因被注释为编码“假定酶”、“小分子运输”、“膜蛋白”、“中心中间代谢”或“能量代谢”、“双组分调控系统”、“分泌因子”、“转录调节因子”和“核苷酸生物合成和代谢”。文献检索表明,已知四个基因hcnABC(编码氰化氢合成酶)和exoY(III型分泌蛋白)在P. aeruginosa的毒力和发病机制中发挥作用。
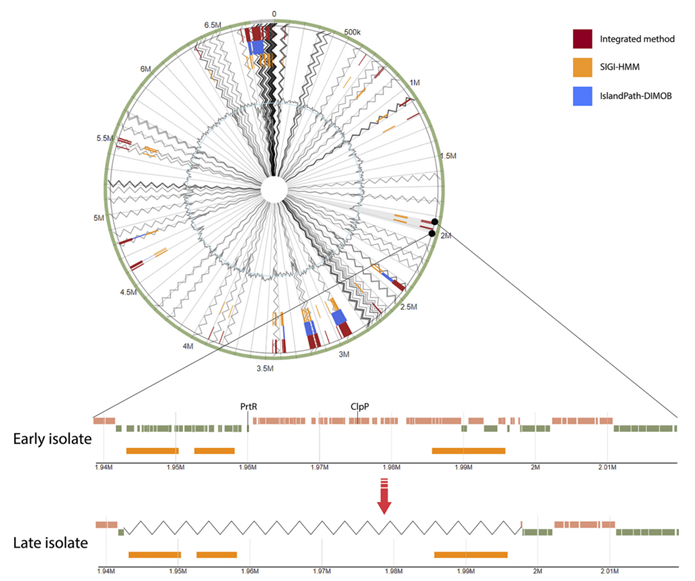
- 前噬菌体相关基因和基因组岛的趋同进化
在52个高变异基因中,有6个被鉴定为来自铜绿假单胞菌噬菌体的前噬菌体origin基因,与假单胞菌噬菌体phi1和phi2的基因类似。且这6个前噬菌体基因包含在variable groups(56 ~ 82个基因)中,可能代表了尚未描述的基因组岛(GIs)(根据基因组岛的特征)。
基于islandviewer4预测,所有六个前噬菌体origin基因都被预测为基因组岛的一部分。此外,在预测的基因组岛中,约90%的基因编码假定蛋白,因此很难确定基因组岛中大部分基因的功能。但预测基因组岛丢失的可能驱动因素可以确定,编码Clp蛋白酶的同源基因或prtR基因,已知是与细菌毒力和致病性相关基因组岛的一部分。
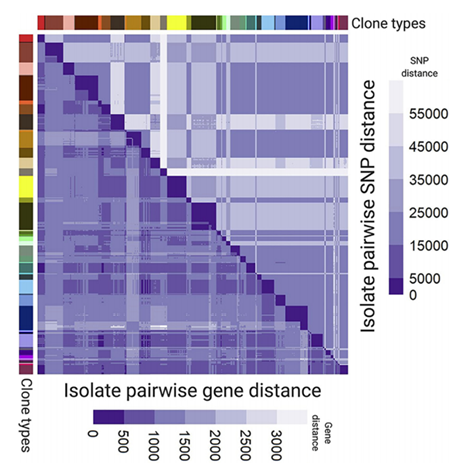
- 总体种群结构:SNPs和基因距离
为确定分离株谱系基因组背景下的总体种群结构,作者根据核心基因组中的SNP(Pactyper)和基因获得丢失(GenAPI)情况确定了446株分离株的遗传关系。基于SNP和基于基因获得丢失的系统发育树都根据克隆类型和患者对分离株进行了聚类。此外,两个系统发育树都显示,这些谱系总体上分别与参考菌株PAO1或UCBPP-PA14聚为一类。
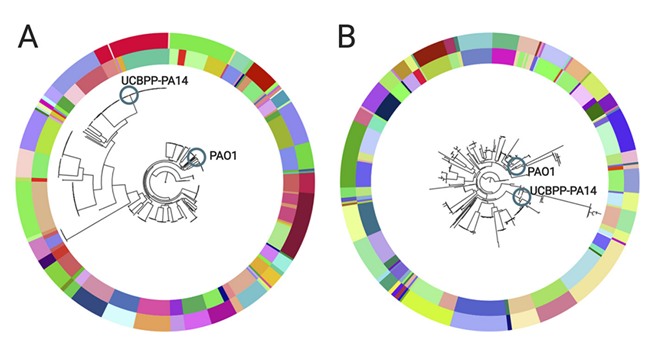
谱系之间的核心基因组SNP成对距离平均为31,909个SNPs,而基因含量差异平均为1,142个基因。PAO1组显示谱系之间的平均多样性为19,853个SNPs和1,043个基因差异;而UCBPP-PA14组平均为35,191个SNPs和1,217个基因差异。Wilcoxon’s rank sum检验发现两组之间存在显著差异,PAO1组内的SNP和基因变异频率低于UCBPP-PA14组。
文章结论
1、铜绿假单胞菌进化过程中,基因丢失的频率远大于基因获得的频率(约6倍);
2、铜绿假单胞菌体内丢失或获得的大多数基因属于基因组中跨谱系非共有的部分(即aggregated accessory genome)。
3、前噬菌体基因是铜绿假单胞菌最常发生丢失或获得的基因,且前噬菌体被认为是新遗传物质的主要来源。
4、质粒的缺少及其可变性可能与铜绿假单胞菌携带质粒的高适应性成本有关。
5、基因丢失通常是以一组基因(基因簇)的形式发生丢失,而不是单个基因。
6、在同一克隆类型的分离株中,每个基因丢失/获得的SNP明显少于来自不同克隆类型的分离株。
7、编码毒力因子的hcnABC、exoY,与前噬菌体相关的Clp蛋白酶基因同源物以及prtR基因是本研究中较为常见的丢失基因。
参考文献
Migle Gabrielaite, Helle K Johansen, Søren Molin, et al. Gene Loss and Acquisition in Lineages of Pseudomonas aeruginosa Evolving in Cystic Fibrosis Patient Airways [J]. mBio, 2020, 11(5): e02359-20.
推荐课程
【课程】微生物比较基因组精品系列课——全套自学必入的系统课程
课程链接:微生物比较基因组精品系列课【全套】
【课程】铜绿假单胞菌基因组研究和分子分型实战
课程链接:铜绿假单胞菌基因组研究和分子分型实战
【课程】微生物比较基因组与群体进化——基因组变异专题研究
课程链接:微生物比较基因组与群体进化
【课程】微生物分子分型-MLST课程——分型全套(含理论、软件、方法)
课程链接:微生物分子分型-MLST课程
【课程】基因组结构分析神器Easyfig实操精品课
【课程】BRIG绘图——结构比较专题2
【课程】肺炎克雷伯菌基因组学研究综合指南
课程链接:肺炎克雷伯菌基因组学研究综合指南
【课程】微生物基因组生信必学课程
课程链接:微生物基因组生信分析必学课程
【课程】微生物生防菌研究
课程链接:生防菌的系统化研究
专题材料
【资料】耐药专题材料
【资料】生防专题材料
请添加唯那生物技术客服小唯的微信号winnerbio01,备注“耐药专题”或“生防专题”,立马获取。
更多专题推荐
CORPORATE CULTURE
1、耐药毒力专题
-
点赞 (0人)
- 收藏 (0人)



