如何绘制这般漂亮的KEGG通路图?
- 看不见的线
- 6338
- 2024-07-01 11:00:16
- 文章来源:基迪奥生物
iPATH 3 (Interactive Pathways Explorer)是一个用于pathway maps分析、作图的交互式在线工具。简而言之,iPATH的主要功能是查找通路和绘制通路,除了可以自由调整图形元素的大小、粗细、颜色等,我个人觉得Time series功能也不错,可绘制出动态的通路图,便于分组数据的比较。
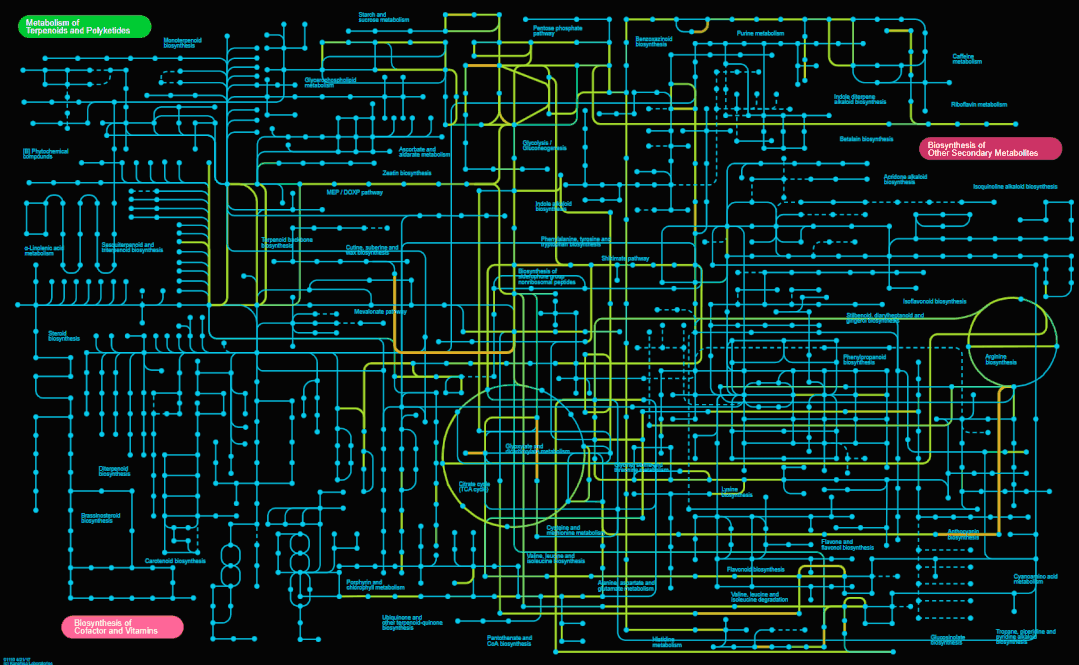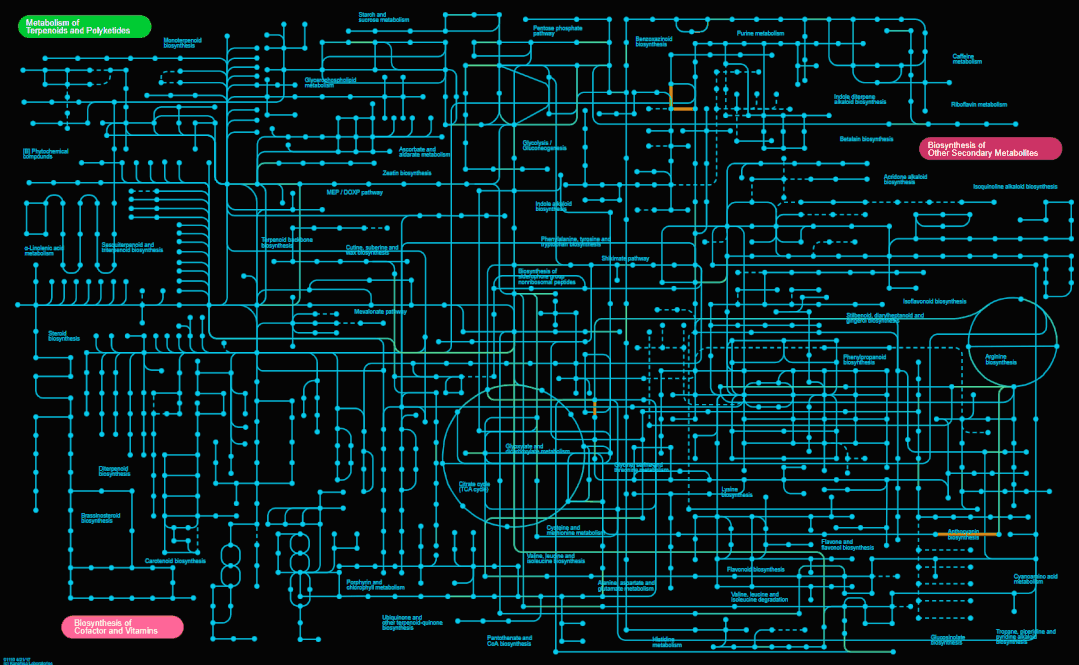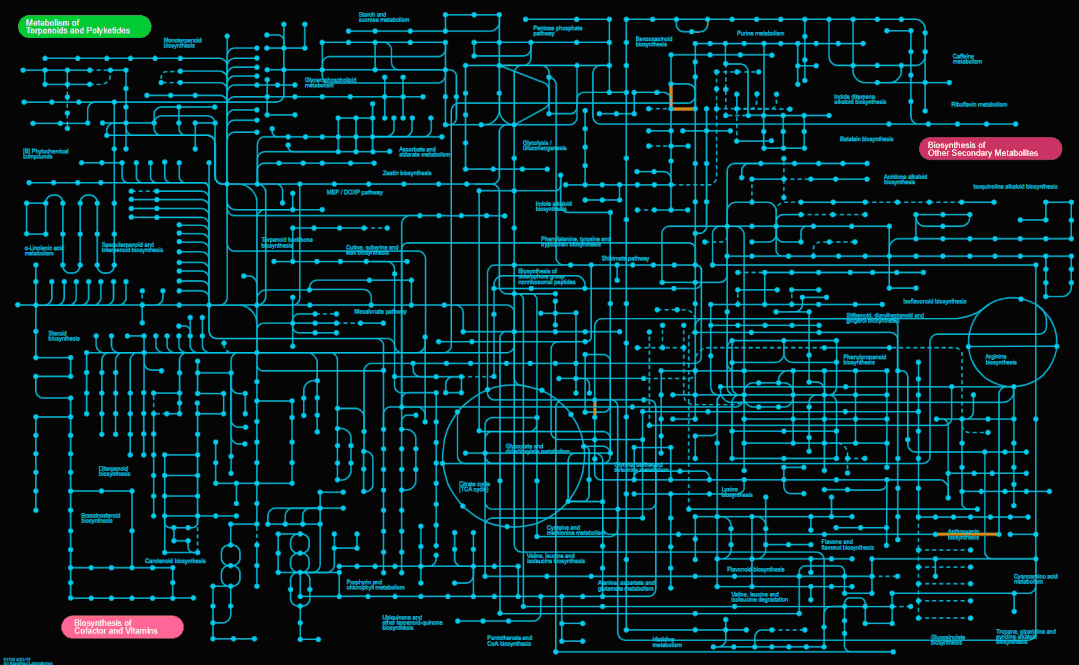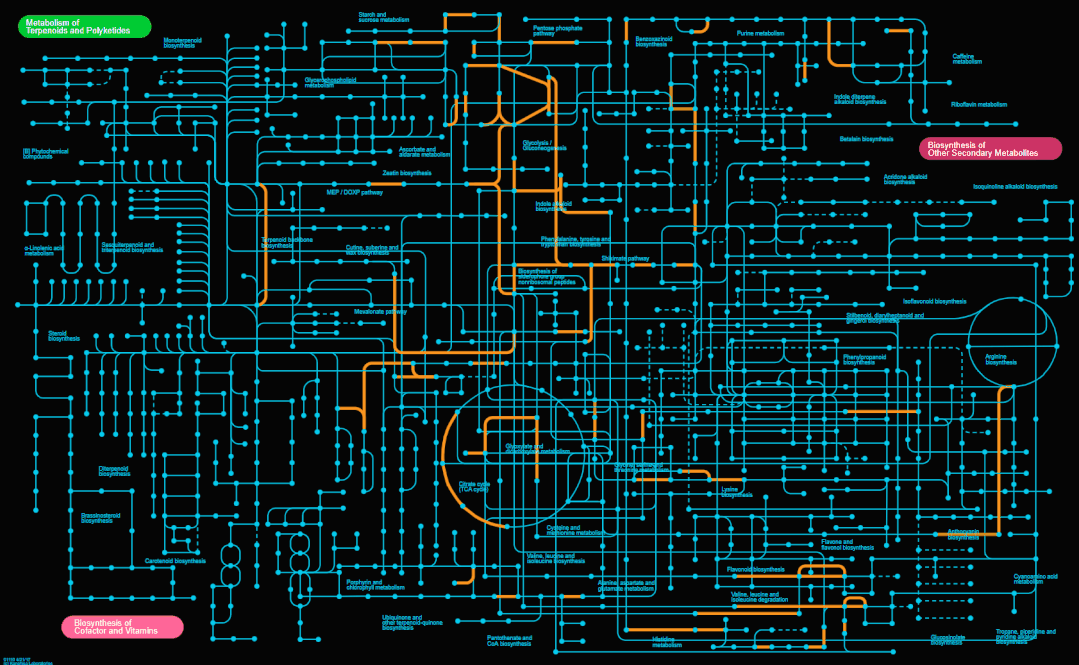
iPATH 3目前提供4张完整的总览图,如下,分别为新陈代谢(Metabolism)、次级代谢产物合成(Secondary metabolites)、抗生素合成(Antibiotics)和微生物代谢(Microbial metabolism)。
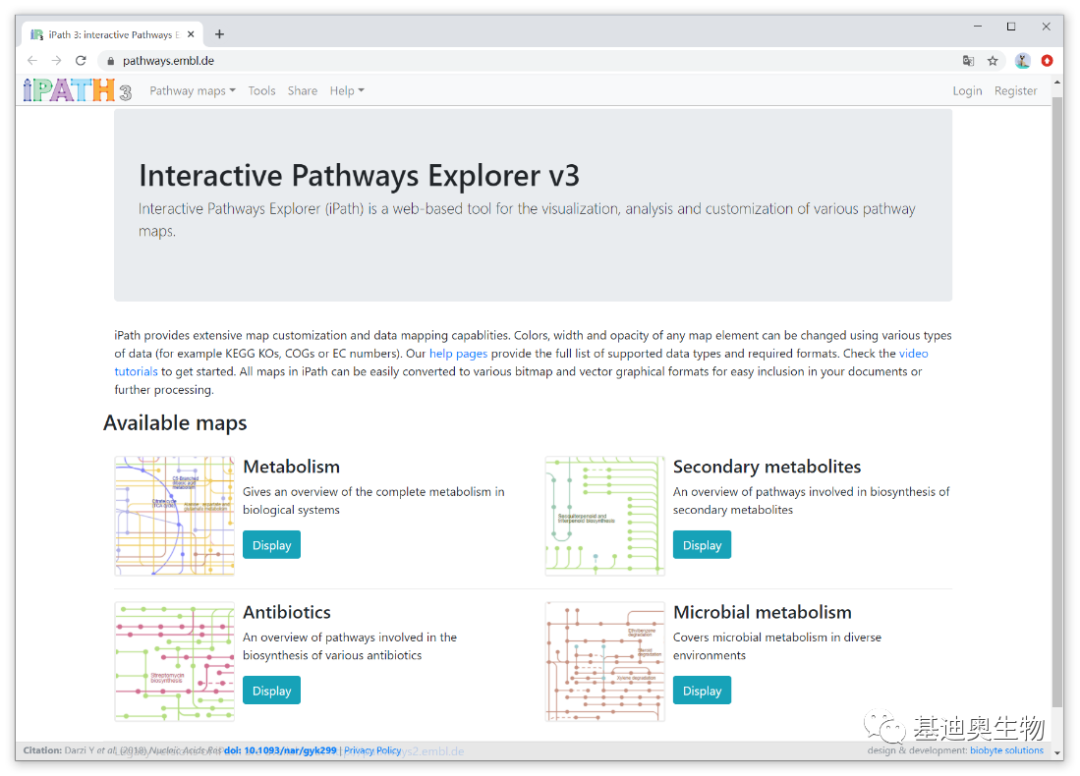
工具网址:
https://pathways.embl.de/
我这里先直接将这4张总览图展示出来,大家一起感受下。
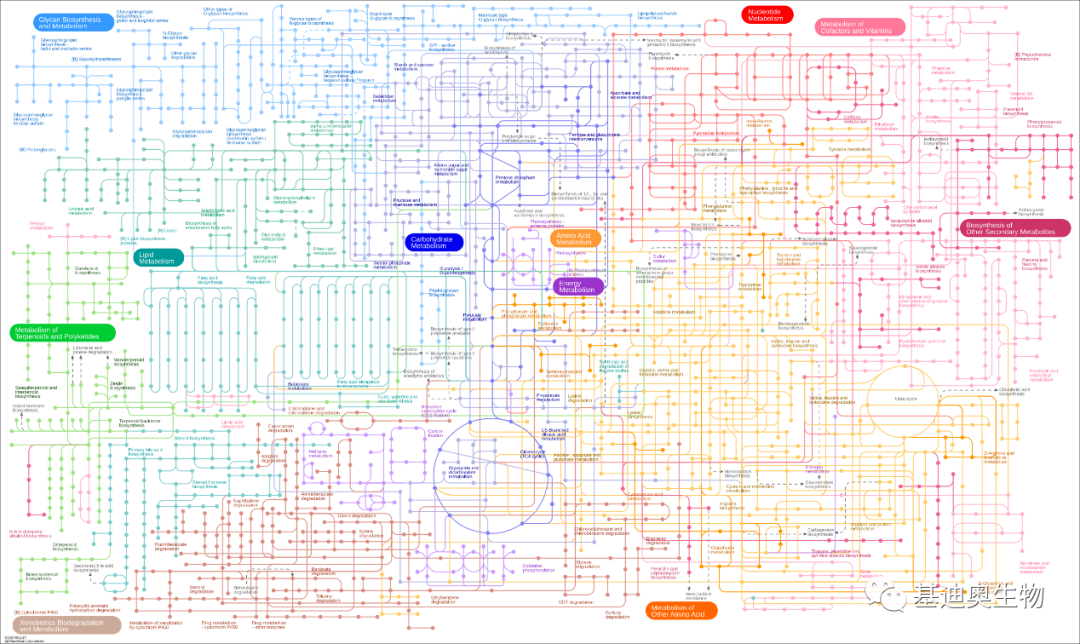
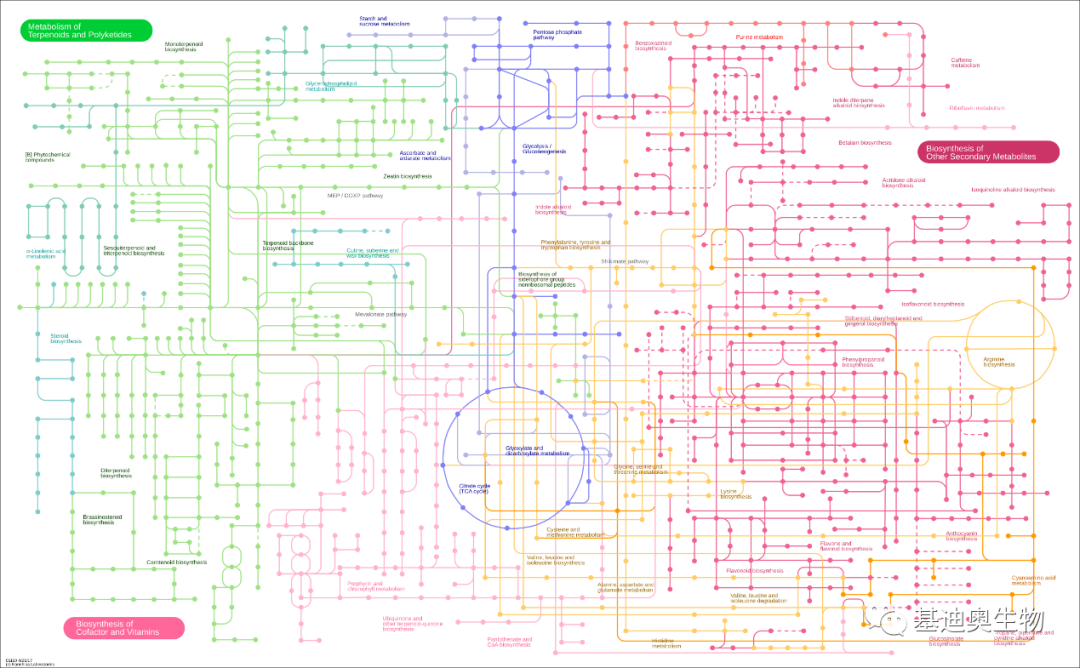
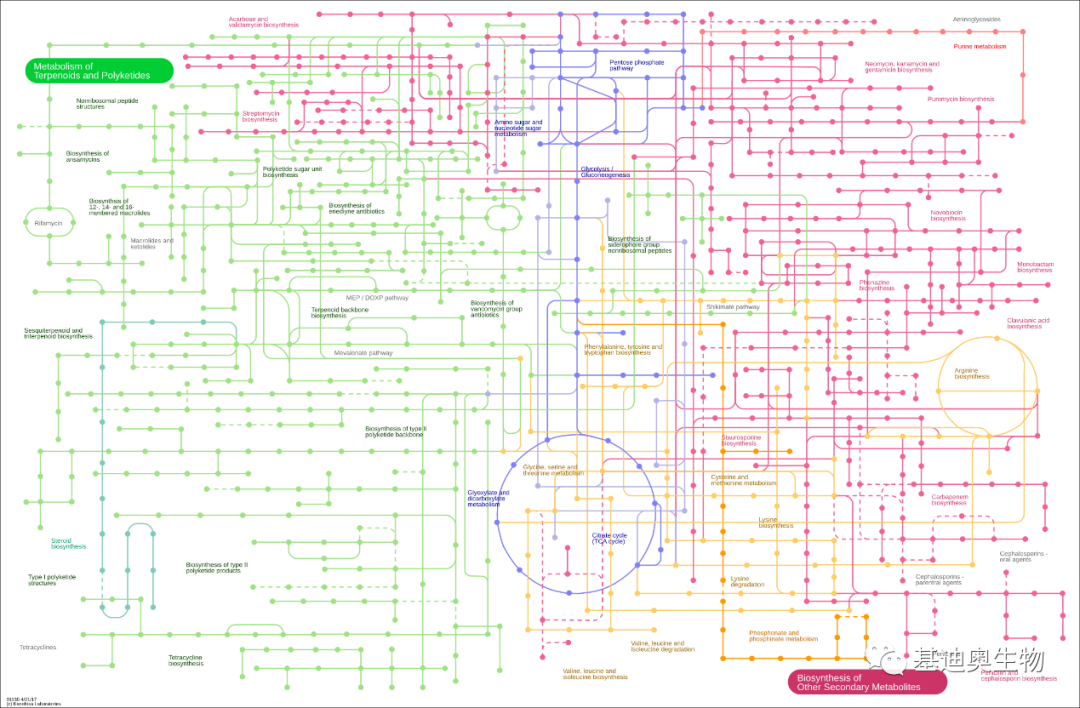
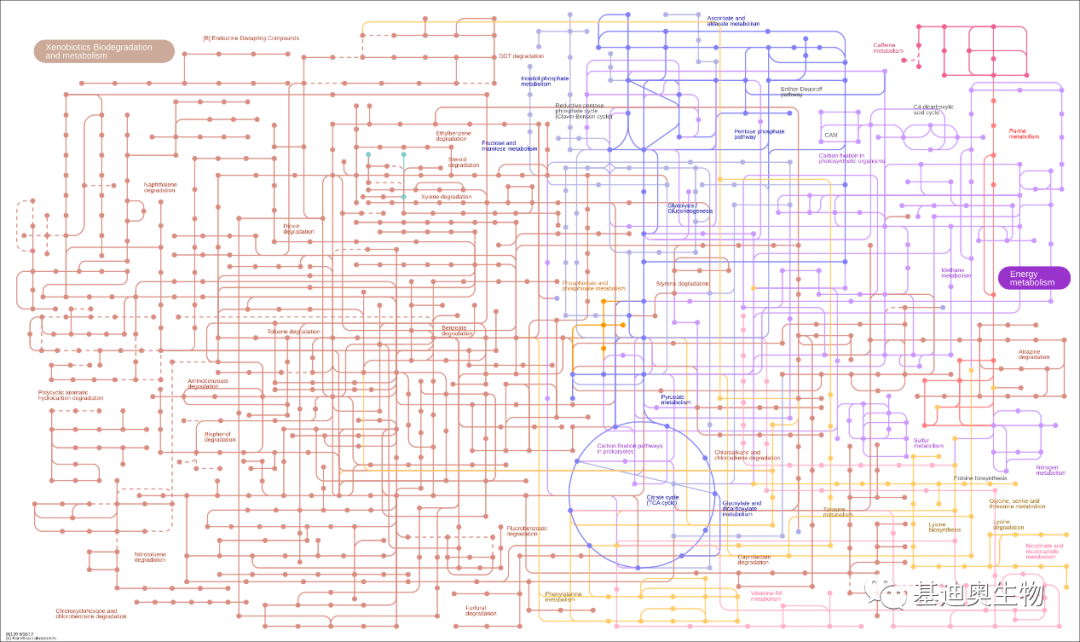
嗯,图非常帅,那么这个工具怎么用呢?
界面介绍
iPath的用户界面主要由图形显示窗口和图形控制窗口两部分构成,如下图。点击窗口左上角的﹢、﹣号按钮,可对视图进行放大缩小,中间的按钮复原。当然,最简单的缩放方式是使用鼠标中间滚轮。点击Pathway maps可在4张maps间切换,右边的Search搜索框可用来搜索感兴趣的pathways、compounds、Enzymes等。
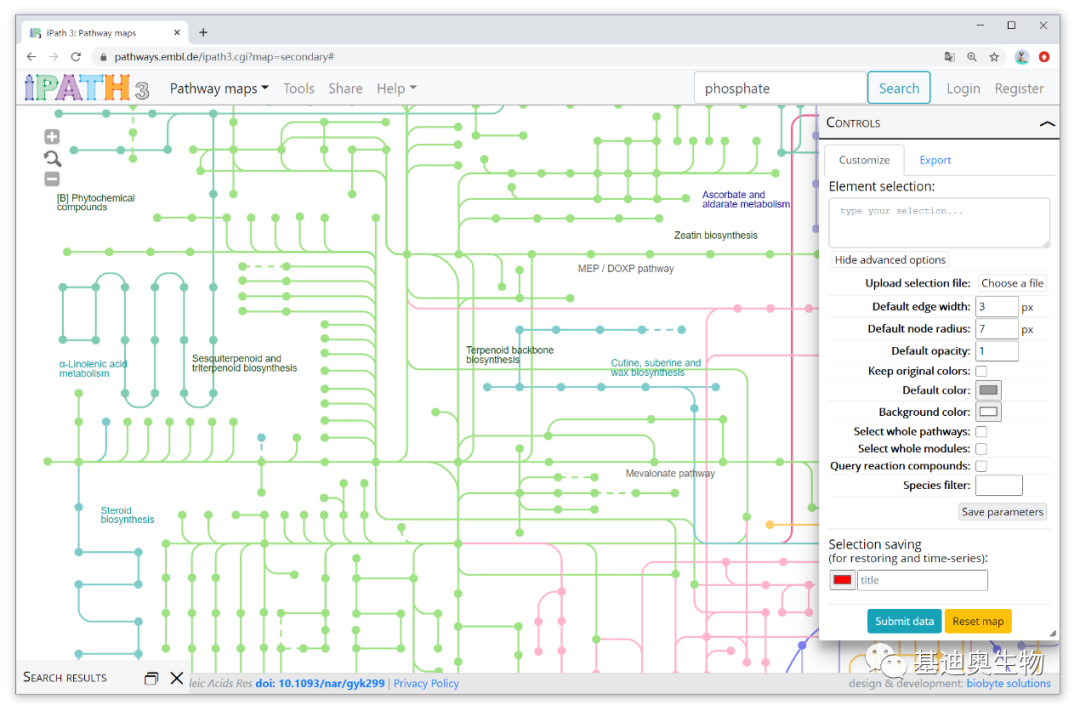
对于页面右侧的Controls窗口,点击Show advanced options按钮,可展开高级设置选项,主要用于设置图形元素的选择方式、大小、颜色等。
查找通路
通过输入“关键词”(如glucose)、化学分子、pathway等的“ID”(如map00030),可在当前选择的map中查找相关联的分子、通路等,查找结果会以表格的方式展示出来。比如我这里输入“phosphate”,会得到以下的结果:
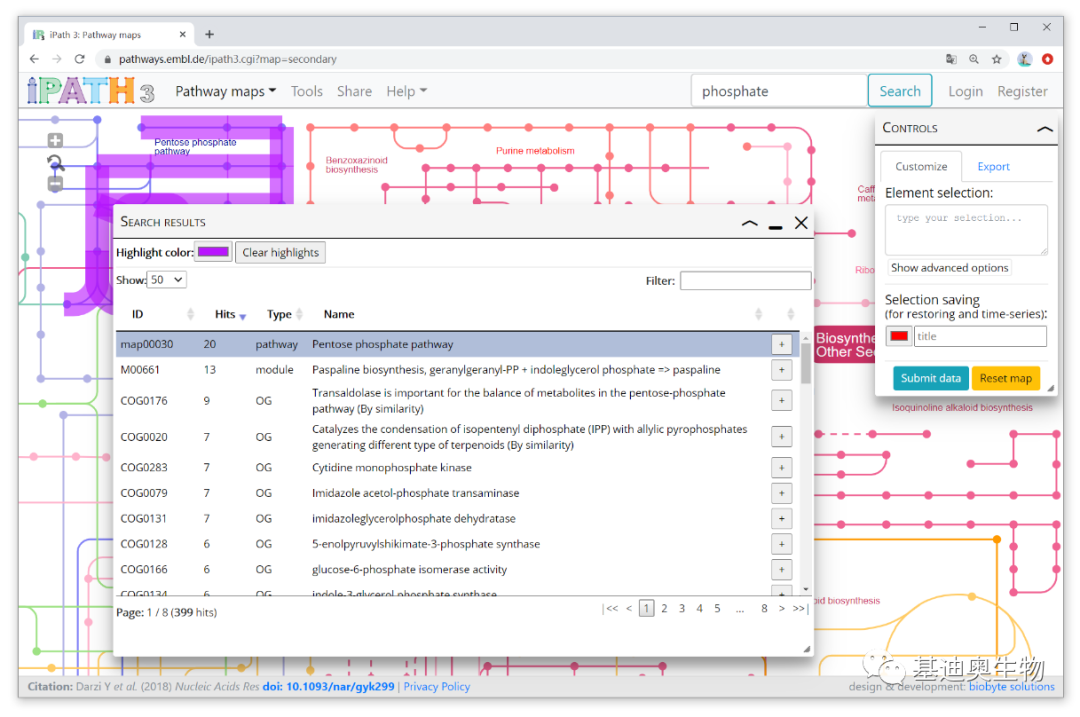
点击图形中的结点或边,就能看到对应的分子、通路信息。点击表格的列名可以对表格按当前列排序,比如我这里点击Hits进行倒序排列。点击查到的每一条记录会在图上高亮显示,点击右侧的加号,会将该条记录的ID添加到CONTROLS窗口的Element selections输入框中,便于进一步个性化绘制目标通路。这里的Type包括Compounds、Pathways、Orthologous groups(OG)、Modules、Enzymes和Reactions六种类型。
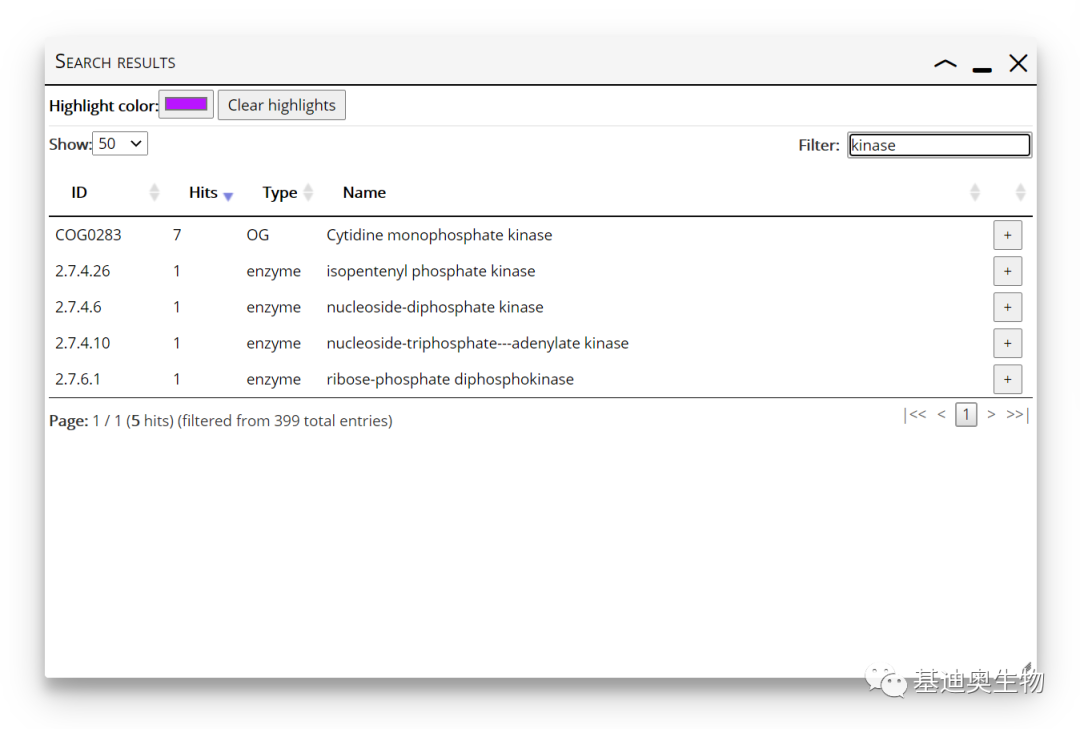
此外,如果搜到的记录非常多,可在Filter输入框中再次添加关键词,进一步过滤查找结果,比如,我这里在Filter中输入“kinase”,结果中只保留Name字段中包含kinase(激酶)的条目。
绘制通路
iPATH本质上已将所有通路画好了,我们所做的其实只需突出展示感兴趣的目标通路(或分子)而已。其中,CONTROLS窗口是我们主要的绘图设置窗口,Element selections输入框是自定义图形元素的主要窗口。通过Element selections,我们可以指定图中突出展示哪些通路、分子,以及图形的颜色、大小(粗细)和透明度。
我这里在Element selections输入框输入以下内容(空格分隔):
M00114 #fc8d1e W5 0.9map00030 #c48dff W5 0.9C00118 W10C00236 W8
这里的第1个字段(列)必须为通路、分子等的id,后面的3个字段(列)可省略(使用默认属性),且没有顺序要求。后面的3个字段主要用来设置图形元素的颜色(Color)、连线的粗细或结点的半径(Width)、图形的透明度(Opacity)。
Color:
可以使用16进制色号、RGB、CMYK色号,例如: #ff0000,RGB(255,0,0) ,CMYK(0,100,100,0) ;
Width:
以 W 为前缀后加数字,单位为px;
Opacity:
0~1之间的数值, 0 表示透明,1 表示不透明。
然后,点击Show advanced options,将Default color调为青色,Background color改为黑色,得到下图这样的效果,Selection match statistics 窗口展示匹配到的条数,点CSV可下载统计结果。
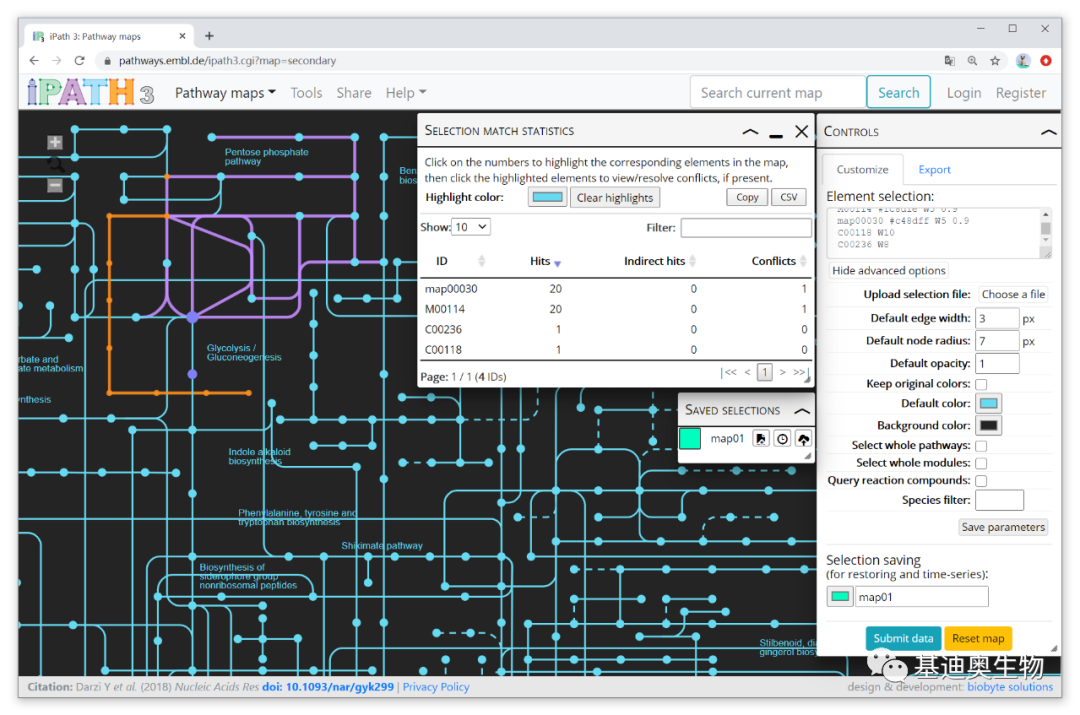
当然,最重要的还是要了解CONTROLS窗口中的这些选项,如下:
Default edge width:
1到30之间的值(以像素为单位),用于指定没有选中边的粗细;
Default node radius:
1到30之间的值(以像素为单位);用于指定没有选中结点的大小;
Default opacity:
介于0到1之间的值,用于指定没有选中图形的透明度;
Keep original colors:
勾选时,map中的所有图形元素都将保持其初始颜色,而选中的图形元素除外;
Default color:
当未勾选“Keep original colors”时,此颜色将用于没有选中的部分;
Background color:
设置背景颜色;
Select whole pathways:
勾选时,会选中与单个边或结点关联的整个pathway;
Select whole modules:
勾选时,会选中与单个边或结点关联的整个KEGG模块;
Query reaction compounds:
当使用KEGG compound id时,勾选后将检查相关联的所有反应中是否存在这些化合物;
Species filter:
使用NCBI taxonomy ID 或KEGG 3 letter species code限定物种,如人的Taxon ID为9606、拟南芥的Taxon ID为3702,仅显示特定生物中存在的map,其余部分将以灰色显示;
Selection title:
填写了标题后,点Submit data时,所有设置被保存并出现在Saved selections窗口中。
除了展示个别感兴趣的分子、通路之外,如果想在通路图中展示所有的差异基因(分子)该如何操作呢?
其实只需拿到基因对应的KO id(大K号)即可,一般测序报告的基因注释总表都有。实在拿不到的话,可以使用OmicShare 的KEGG功能注释工具重新注释。
工具链接:
https://www.omicshare.com/tools/Home/Soft/kegg_anno
使用Excel将基因的KO id(必须为第1列)、连线的粗细/结点的半径(Width)以及色号整理成3列,如下,然后另存为制表符分隔的文本文件(txt)。
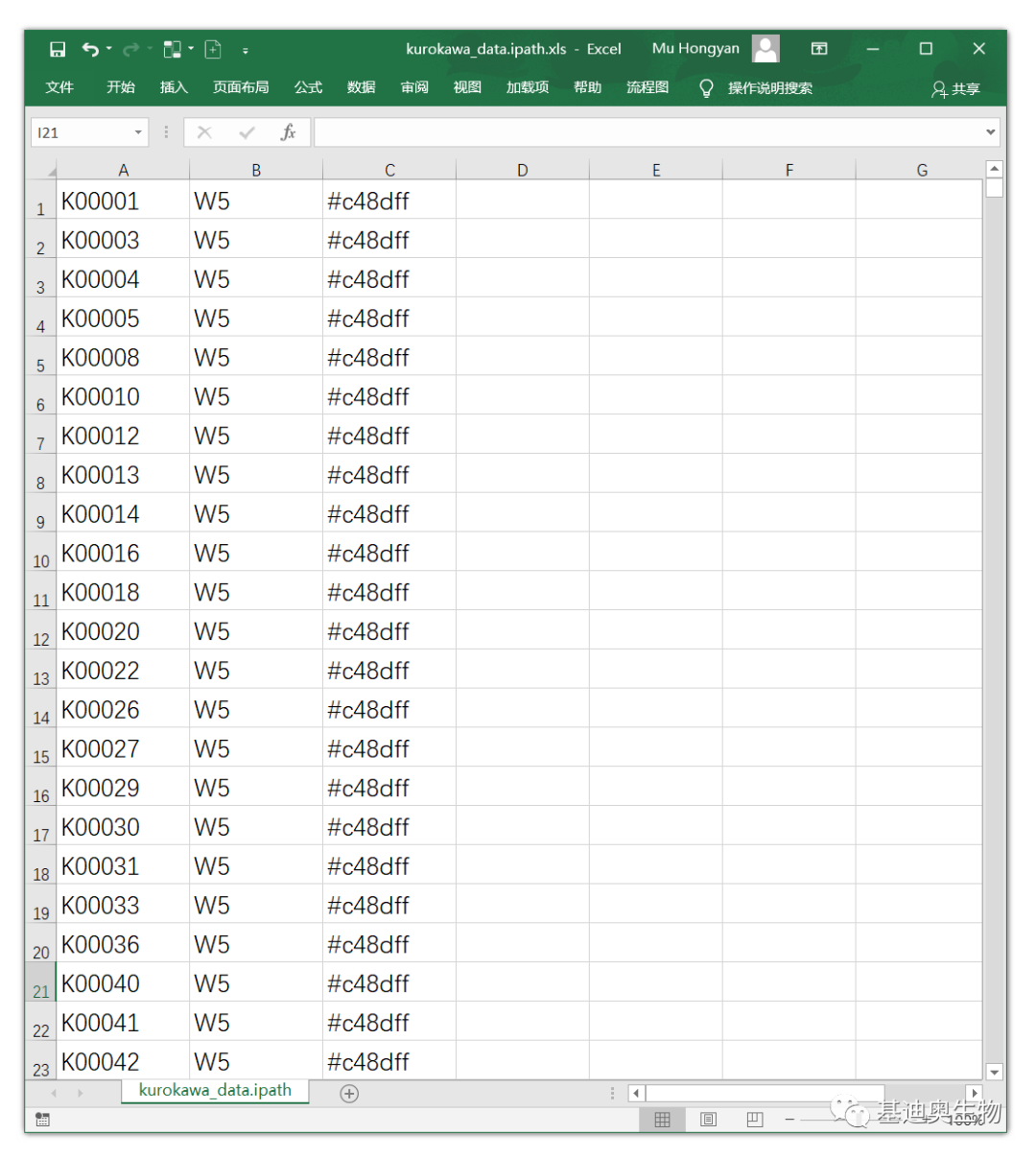
注意,宽度值前需要加上W,W后的数值可以不同,实现类似网络图那般连线粗细不一的效果。如果想设置透明度也再加1列。为了方便大家练习,本文用到的范例数据已上传到OmicShare 论坛上,感兴趣的小伙伴可以前去下载。
下载链接:
https://www.omicshare.com/forum/thread-6927-1-1.html
数据保存好后无需将制表符替换成空格,直接在CONTROLS窗口,点击Show advanced options,然后通过Upload selection file,上传刚才准备好的制表符分隔的文本文件,然后点击Submit data按钮即可完成绘制,效果如下。
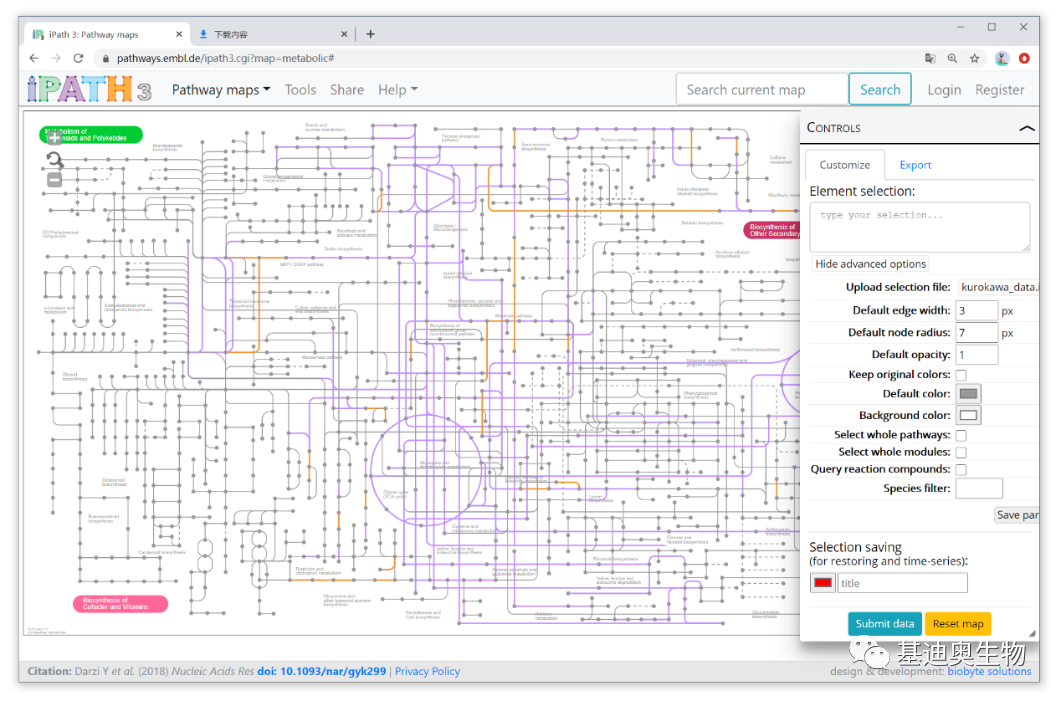
我这里没有调整其他参数,Pathway maps选择Secondary metabolites,其他一切保持默认。为了突出显示指定基因所在的通路,这里的Default color为灰色,如果主要以电子版形式展示,也可以将背景颜色(Background color)改为黑色,这样更炫酷一些!
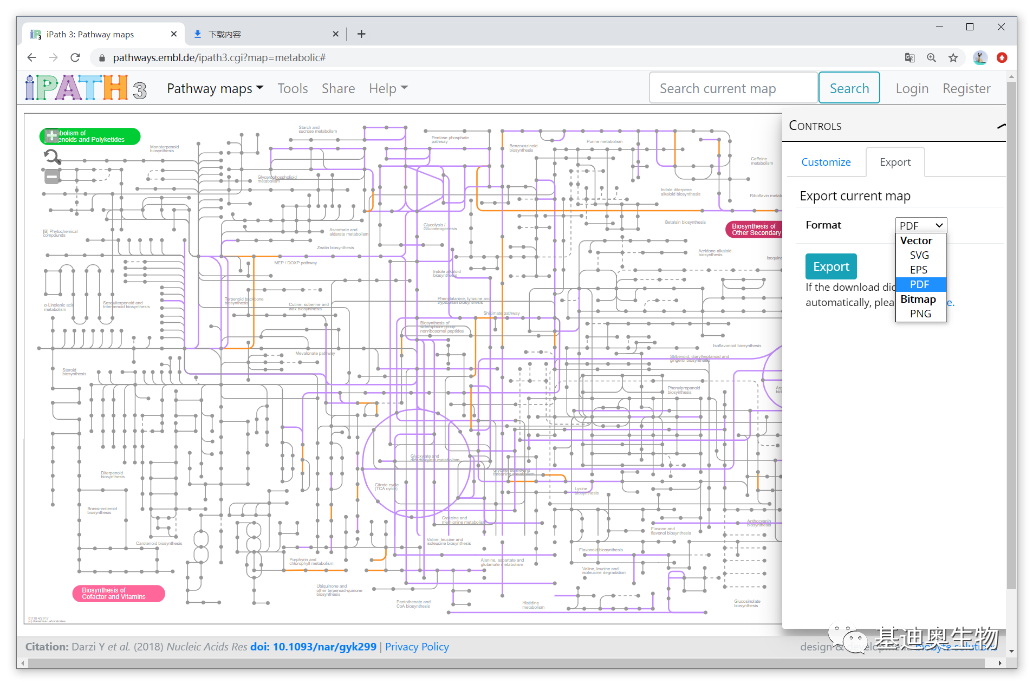
满意后,可以保存为矢量图形格式(比如常见的pdf和svg)以便于做进一步的后期处理。例如,想给部分pathway来个特写,可使用Adobe illustrator软件打开svg格式的矢量图,对感兴趣的区域创建剪贴蒙版。我这里推荐保存为svg格式,这样Ai更容易识别字体。
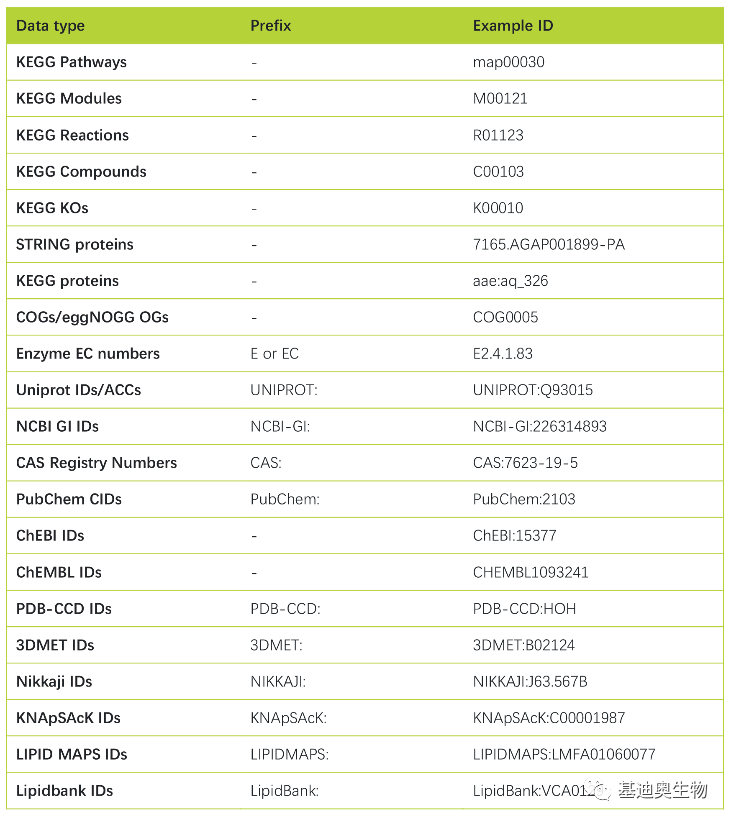
此外, iPath提供强大的pathway maps绘制和数据映射功能,支持各种类型的数据id(例如KEGG KOs、COGs 、EC numbers等,见下表),不过,注意使用时加上前缀。
表1iPath支持的id类型
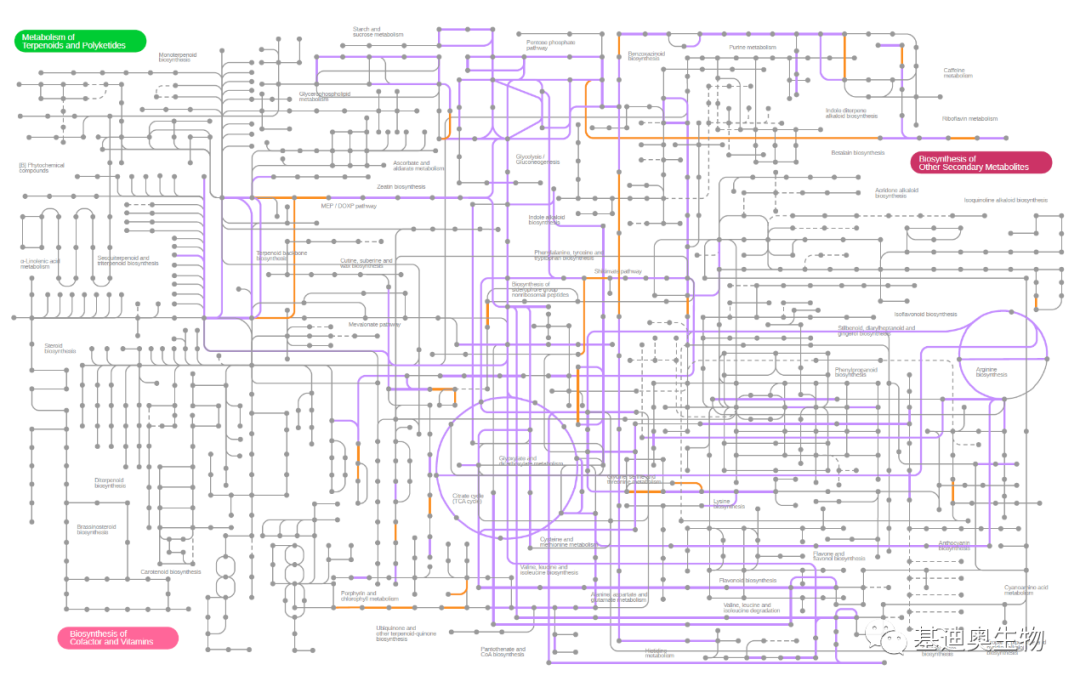
最终绘制的效果如下:
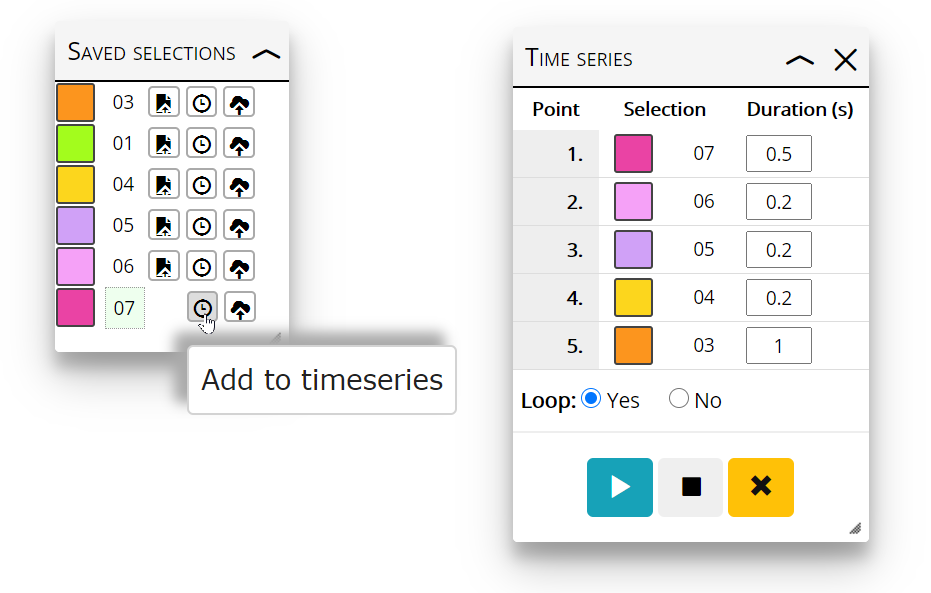
当然,我们也可以将保存的多个map添加到时间轴窗口,方法如下图,设置每一帧画面持续的时间(单位秒),勾选Loop(循环)后,点下方的播放按钮即可实现播放动画。
结合其他录频软件即可生成短视频或动图,效果如下。
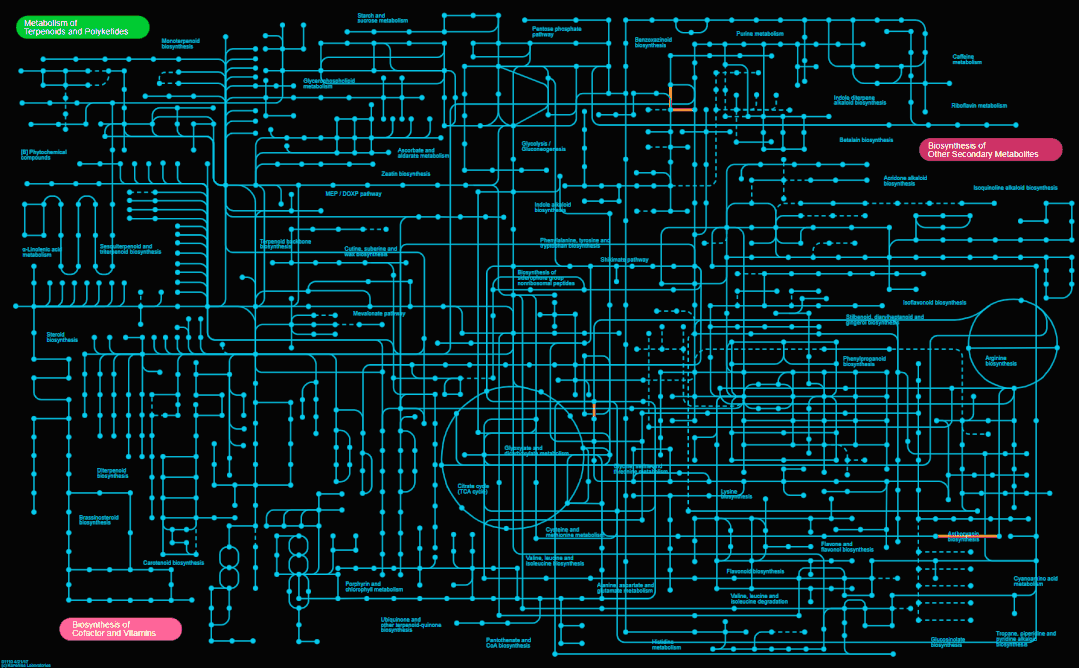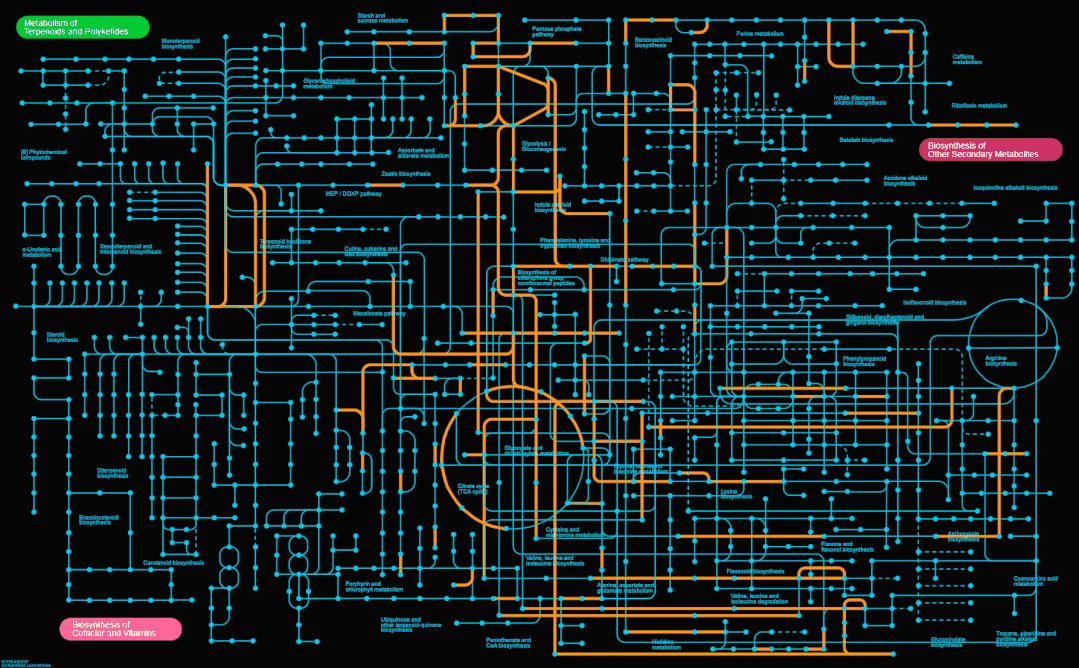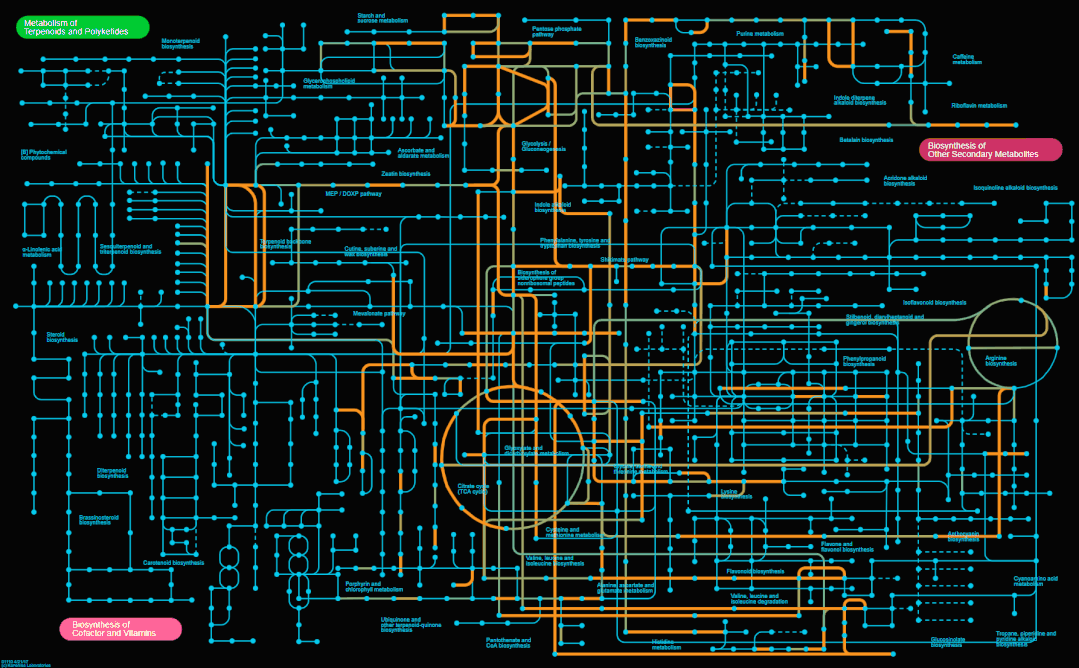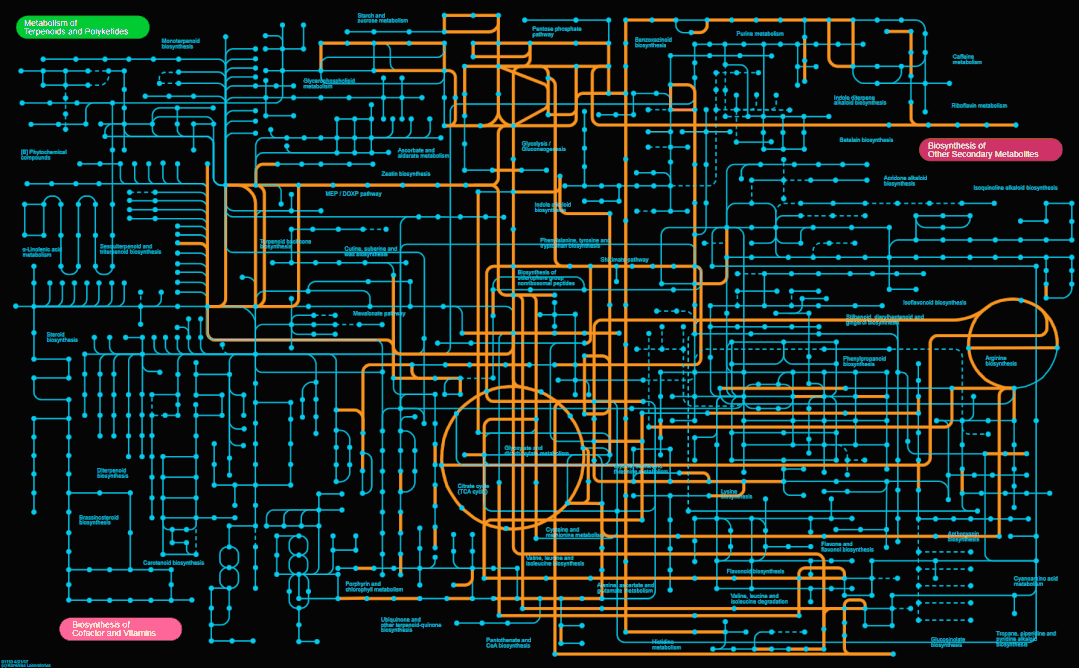
最后,我个人觉得ipath绘制的通路图和Cytoscape、Gephi等软件绘制的网络图类似,主要呈现出一种组学研究整体的、全局的描述,而不是具体的一两个基因。好啦,今天就分享到这里啦~
-
点赞 (0人)
- 收藏 (0人)



