实用组学小工具——大肠埃希菌亚群分类和鉴定
- 看不见的线
- 736
- 2025-09-05 10:57:10
- 原创
一、大肠埃希菌亚群介绍
埃希氏菌属(Escherichia)包括艾伯特埃希菌(E. albertii)、弗格森埃希菌(E. fergusonii)、五个埃希菌隐蔽进化枝以及严格意义上的大肠埃希菌(E. coli sensu stricto)。其中大肠埃希菌可进一步划分为七个主要系统群(phylogroup),分别命名为A、B1、B2、C、D、E和F。由于这些物种/系统群具有特定的生存方式和宿主偏好性,它们的鉴定对流行病学研究具有重要意义。
传统表型检测法无法准确识别非严格意义大肠埃希菌及各类系统群。Clermont及其团队开发的PCR检测方法实现了对大多数物种/系统群的鉴定,其中三重/四重PCR技术已成为大肠埃希菌系统群分型最常用的方法。随着全基因组测序技术的普及,研究者开发了ClermonTyping方法及其网络交互界面ClermonTyper。该工具能够将菌株序列精准归类至艾伯特埃希菌、弗格森埃希菌、埃希菌隐蔽进化枝(I-V)和严格意义上的大肠埃希菌phylogroup(A-F)。
ClermonTyping延续了体外PCR检测的设计理念,保持了操作简便和快速高效的特点。该计算机分析方法与体外PCR检测结果的吻合度达99.4%,与Mash基因组聚类工具的一致性达98.8%。极少数误差主要源于水平基因转移或引物中的单核苷酸多态性(SNP)。
二、大肠埃希菌亚群分类和鉴定小工具
密码子实验室在生信云平台中添加了大肠埃希菌亚群分类和鉴定(ClermonTyping)小工具:用户上传fasta格式的菌株全基因组序列文件或包含多个目标基因组的文件夹(输入文件后缀为 *.fasta/*.fa),即可实现大肠埃希菌亚群分类和鉴定。
地址:https://cloud.mimazi.net/tool/article-197.html
版本:Clermont Typing Current version : 24.02 (Fev. 2024)
使用方法:
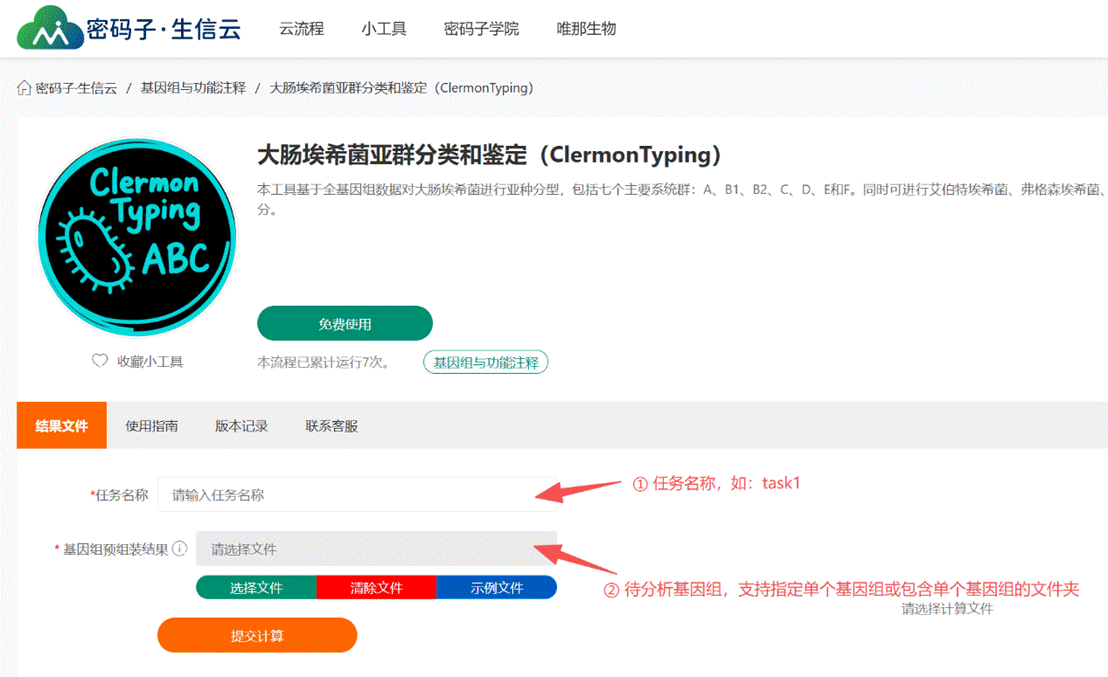
输入文件:
基因组预组装结果(FASTA格式),支持提交单个基因组或包含多个目标基因组的文件夹,输入文件后缀为 *.fasta/*.fa
文件名禁止包含空格(如:EC Strain1.fasta,可将其修改为 EC_Strain1.fasta)
输出结果:
clermonTyping_phylogroups.txt:ClermonTyping的最终输出

第一列:被分析样品的名称;
第二列:PCR检测到的所有基因(包括可能无用的验证基因);
第三列:在四重列中,目标基因(arpA, chuA, yjaA,TspE4.C2)的存在(+)/缺失(-)表示;
第四列:当四重PCR结果不明确时,该列显示C组和E组的特异性等位基因;
第五列:关于系统群的最终结论。
三、密码子生信云带您零基础做生信
密码子·生信云平台优势
零门槛体验:无需本地安装Linux环境,无需复杂配置,打开网页即可一键上传数据,轻松运行各种分析。
高效安全:云端计算资源强大,数据处理速度快,保障数据安全与隐私。
友好界面:操作界面简洁直观,适合各类用户,无需编程基础。
专业支持:提供详细的操作指南和技术支持,助力您的科研工作顺利进行。
密码子生信云还提供云流程和其它各色小工具分析服务:
为细菌基因组研究量身定制的智能云流程从基因组测序原始数据(fastq)或组装序列(fasta)出发,一键化完成细菌基因组全套分析流程,包括基因预测、多功能注释和多数据库分析。多种多样的实用小工具,可以为您精准解决碎片化需求,包括序列处理、统计绘图、基因组注释等等,助力您轻松完成数据分析。
参考文献:
-
点赞 (0人)
- 收藏 (0人)



