BLAST本地比对操作指南
- 看不见的线
- 1297
- 2025-06-06 08:57:25
- 原创
BLAST(Basic Local Alignment Search Tool)是一款由 NCBI(美国国家生物技术信息中心)提供的生物信息学工具,用于比较基因或蛋白质序列。以下是如何在本地使用 BLAST工具进行核酸或蛋白序列比对。
一、常用 BLAST子模块简介
| 子模块名称 | 查询序列类型 | 数据库序列类型 | 比对级别类型 |
| blastn | 核酸 | 核酸 | 核酸 |
| blastp | 蛋白 | 蛋白 | 蛋白 |
| blastx | 核酸 | 蛋白 | 蛋白 |
| tblastn | 蛋白 | 核酸 | 蛋白 |
| tblastx | 核酸 | 核酸 | 蛋白 |
- 二、使用BLAST软件构建本地数据库(核酸数据库和蛋白数据库)
BLAST数据库分为两类,核酸数据库和氨基酸数据库,可以用makeblastdb创建。
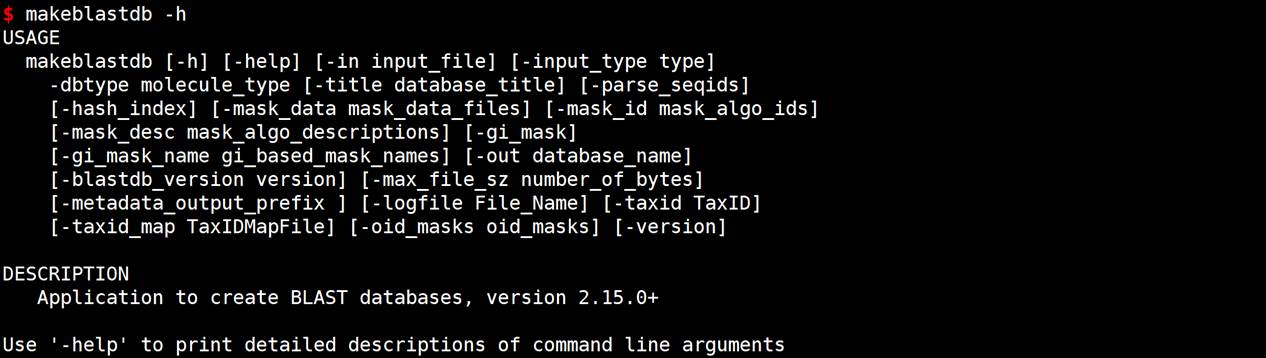
#常用参数说明:
- -dbtype是必选参数,nucl, prot,二选一,核酸序列或者氨基酸序列库
- -in用于构建搜索库的fasta文件
- -input_type默认为fasta文件,其他支持文件asn1_bin,asn1_txt, blastdb
- -out输出文件的前缀
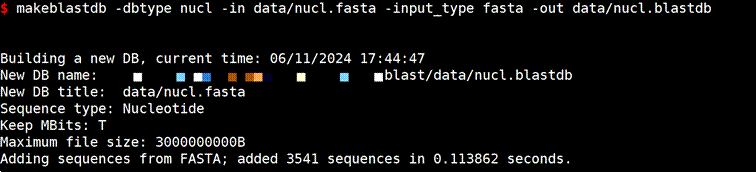
#创建核酸数据库
makeblastdb -dbtype nucl-in data/nucl.fasta -input_type fasta -out data/nucl.blastdb
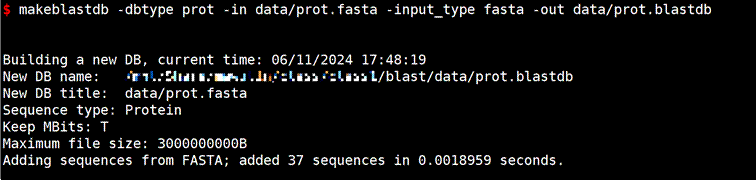
#创建蛋白数据库
makeblastdb -dbtype prot-in data/prot.fasta -input_type fasta -out data/prot.blastdb
- 三、使用BLAST软件进行常规比对
3.1将核酸序列比对至核酸数据库(blastn)
blastn-query data/nucl_query.fasta -out data/blastn_nucl_result.tsv -db data/nucl.blastdb -num_threads 16 -evalue 1e-10 -max_target_seqs 5 -outfmt 6
3.2 将蛋白序列比对至蛋白数据库(blastp)
blastp-query data/prot_query.fasta -out data/blastp_prot_result.tsv -db data/prot.blastdb -num_threads 16 -evalue 1e-10 -max_target_seqs 5 -outfmt 6
3.3将核酸序列比对至蛋白数据库(blastx)
blastx-query data/nucl_query.fasta -out data/blastx_nucl2prot_result.tsv -db data/prot.blastdb -num_threads 16 -evalue 1e-10 -max_target_seqs 5 -outfmt 6
3.4将蛋白序列比对至核酸数据库(tblastn)
#蛋白-核酸,比对时,将输入的氨基酸序列与数据库中核苷酸序列翻译后的氨基酸序列逐一比对
tblastn-query data/prot_query.fasta -out data/blastx_prot2nucl_result.tsv -db data/nucl.blastdb -num_threads 16 -evalue 1e-10 -max_target_seqs 5 -outfmt 6
3.5常用参数介绍
| -query | 指定要用作查询的序列文件 |
| -out | 指定比对结果输出路径 |
| -db | 指定比对的数据库路径 |
| -num_threads | 指定使用的线程数,以加快比对的速度 |
| -evalue | 设置E值阈值,低于1e-5就可认为序列具有较高的同源性 |
| -max_target_seqs | 设定最大保留匹配序列数 |
| -outfmt | 若设置输出格式为6,即以制表符格式输出比对结果(官方提供的输出格式有19种:0-18) |
推荐课程
-
点赞 (0人)
- 收藏 (0人)



