RPS-BLAST使用
- 看不见的线
- 29
- 2025-07-16 08:30:28
- 原创
RPS-BLAST类似于 HMMER,可用于在CDD数据库里搜索某蛋白是否含有一domain或者是否属于某一family寻找蛋白序列具有的保守Domain。
蛋白序列的Domain对于蛋白质来说是最重要的一部分,一般来说会有发挥功能的区域,结合其它物质的区域,二聚化区域等等。
RPS-Blast安装和使用:
# 数据下载,这个文件是包含了NCBI网页版的各个库的,即CDD、COG、SMART、TIGR、PRK等。
wget https://ftp.ncbi.nih.gov/pub/mmdb/cdd/cdd.tar.gz
# 解压
tar -xzvf cdd.tar.gz
# 格式化数据库
makeprofiledb -title Pfam.v.xxx -in Pfam.pn -out Pfam -threshold 9.82 -scale 100.0 -dbtype rps -index true
# -title是给输入的文件的命名(可有可无)
# -in是输入文件,即需要被制作数据库的文件,pn文件中存放的是smp文件名
# -out为输出文件,即数据库的名字,后续使用此名字进行rpsblast
# -threshold向查找表中添加词条的最小分数,默认值为 9.82(Minimum word score to add a word to the lookup table)
# -scale PSSM缩放因子。默认值为 100
# -dbtype输出数据库类型。可以是 cobalt、delta或 rps,默认值为 rps
# -index是否产生索引文件,默认值为true

# 运行搜索
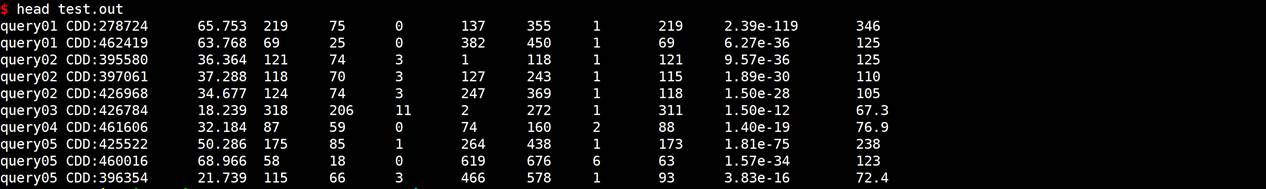
rpsblast -query data/prot_query.fasta -outfmt 6 -evalue 1E-10 -out test.out -db Pfam
# -query为查询序列,fasta格式
# -outfmt输出格式,跟其它blast格式一样,有很多种选择,与其它blast结果类似,参考rpsblast -help
# -evalue自己设置evalue阈值,但是一般Domain的evalue都挺低的
# -out输出文件
# -db使用的数据库,是之前建好的
推荐课程
-
点赞 (0人)
- 收藏 (0人)
-
上一篇: 没有了
- 下一篇: Easyfig罕见问题1



