实用组学小工具——多序列比对可视化展示
- 看不见的线
- 750
- 2025-08-14 09:53:26
- 原创
一、什么是多序列比对?
多序列比对(Multiple Sequence Alignment, MSA)是一种将多个生物学序列(如DNA、RNA或蛋白质序列)进行比对排列,以识别它们之间相似性和差异性的生物信息学方法。
在比对过程中,多个序列中相同或相似的部分会尽可能被对齐到同一列,而插入或缺失的区域则以“-”进行表示。

二、多序列比对与BLAST比对的差异
多序列比对(MSA) | BLAST比对 | |
比对类型 | 全局比对,适用于≥3条序列 | 局部比对,适用于2条序列 |
主要目的 | 分析序列整体相似性 推断进化关系 | 快速匹配相似序列 查找局部相似片段 |
结果形式 | 序列对齐矩阵 | 相似性统计表格 |
典型应用 | 系统发育分析 保守位点鉴定 | 同源基因搜索 功能注释 |
三、多序列比对结果的展示方法
ESPript 3.0(https://espript.ibcp.fr/ESPript/cgi-bin/ESPript.cgi)是一款适用于展示多序列比对结果的工具,能够清晰呈现同源序列之间的整体相似性和细微差异。此外,它还可以标注蛋白质的二级结构,帮助分析突变位点与二级结构的关系。
使用方法:
1、生成对齐序列结果(fasta格式),比如使用MEGA等软件的ClustalW或MUSCLE功能进行多序列比对。
2、将比对文件上传至ESPript 3.0网页。
示例:
以mcr-9.1~mcr-9.4的多序列比对结果为例,由于序列较长,这里仅展示后半部分差异区域的情况。

四、密码子生信云带您零基础做生信
如果需要忽略保守区域,突出不同序列之间的SNP差异,可以尝试上海唯那生物生信云平台(https://cloud.mimazi.net)的“多序列比对可视化(snipit)”小工具。上海唯那生物密码子生信云将操作流程简单化,无需安装软件,无需配置环境,即可快速输出可视化图片(pdf格式)。
您仅需微信扫码注册一个账号,找到免费小工具“多序列比对可视化(snipit)”,提交多序列比对文件(fasta格式),并选择所分析序列的类型(核酸/蛋白),就能快速得到绘图结果。
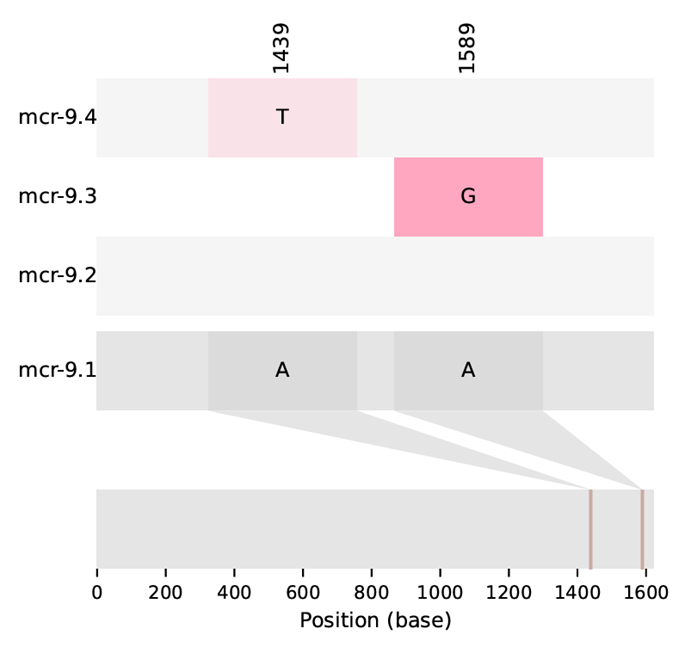
需要注意的是该程序默认以文件中的第一条序列作为参考序列。如下图所示,SNP差异位点1439和1589指向的是差异碱基在mcr-9.1上的位置。同时Indel并不会显示在图中。
以下两项工具同样基于多序列比对数据进行分析,可根据需求选择使用:
“基于多序列比对结果分析SNP/Indel”(http://cloud2.mimazi.net:9001/tool/article-167.html)
“基于多序列比对文件生成SNP距离矩阵(snp-dists)”(http://cloud2.mimazi.net:9001/tool/article-152.html)
此外,密码子生信云还提供云流程和其它各色小工具分析服务。
为细菌基因组研究量身定制的智能云流程从基因组测序原始数据(fastq)或组装序列(fasta)出发,一键化完成细菌基因组全套分析流程,包括基因预测、多功能注释和多数据库分析。而多种多样的实用小工具,可以为您精准解决碎片化需求,包括序列处理、统计绘图、基因组注释等等,助力您轻松完成数据分析。

五、结语
在微生物研究领域,生物信息学分析已不再是专业生信人员的"特权"。密码子生信云平台如同您的随身生信助理,能够将晦涩的技术流程转化为网页端的简单点击。无论您是初次接触生信的临床研究者,还是专注微生物研究的科研工作者,这里都为您准备了零成本的分析入口和零门槛的操作体验。
现在访问平台,开启您的第一个生信分析项目吧!我们始终相信,技术的价值在于让每个人都能平等地获取科研工具,而您要做的,只是保持探索的好奇心。
-
点赞 (0人)
- 收藏 (0人)



