实用组学小工具——大肠埃希菌血清型分析SerotypeFinder和ECTyper
- 看不见的线
- 27
- 2025-09-05 11:00:18
- 原创
一、关于大肠埃希菌的血清型鉴定
大肠埃希氏菌的抗原结构主要包括四类:菌体抗原(O抗原)、表面抗原(K抗原)、鞭毛抗原(H抗原)和菌毛抗原(F抗原)。
在该菌的血清分型中,通常以O抗原和H抗原作为主要分型依据,例如常见的O157:H7型。对于部分致病菌株,还需额外鉴定K抗原,例如O18:K1:H7。
目前,O抗原和H抗原的鉴定通常可借助两种基于全基因组的血清型分析工具——SeroTypeFinder和ECTyper。其中,SerotypeFinder(https://cge.food.dtu.dk/services/SerotypeFinder/)由Joensen等学者开发,通过识别O抗原基因(如wzx、wzy、wzm、wzt)和H抗原基因(如fliC、flkA、fllA、flmA、flnA)来预测血清型,其结果与传统血清学实验高度一致。
ECTyper(https://github.com/phac-nml/ecoli_serotyping)是另一款集成化分析工具,能够基于全基因组测序(WGS)对大肠埃希菌进行多项表征,包括物种鉴定、O/H血清分型、志贺毒素分型以及致泻性大肠杆菌(DEC)病理分型。研究表明,在一项涵盖6954个公开的大肠埃希菌基因组的大规模比对中,ECTyper与SerotypeFinder在O抗原和H抗原分型上的一致性分别达到91%和90%,显示出高度吻合的分析结果。
需要注意的是,基因组流行病学中心(CGE)的SeroTypeFinder工具每次仅支持单个菌株的O/H血清型分析;ECTyper支持批量分析,但需要在本地安装部署,对缺乏生物信息学背景的研究人员而言可能存在一定技术门槛。
另外,K抗原的鉴定目前仍需依靠特异性PCR方法,例如针对neuC(K1)和kfiC(K5)等基因的检测,这类方法可有效补充全基因组分型工具尚未完全覆盖的抗原类型。
二、密码子生信云带您零基础做生信
上海唯那生物推出的生信云平台服务(https://cloud.mimazi.net)提供了上述两种大肠埃希菌血清型预测的小工具。用户无需安装软件或配置环境,即可便捷地完成大肠埃希菌血清型分析,快速获取血清型预测结果(表格形式)。

您仅需微信扫码注册一个账号,找到免费小工具“大肠杆菌分离株血清型预测(SerotypeFinder)”或“大肠杆菌血清分型、志贺毒素分型和病理分型(ECTyper)”,提交对应文件,就能快速预测并下载结果。
小工具地址:
- 大肠杆菌分离株血清型预测(SerotypeFinder):https://cloud.mimazi.net/tool/article-195.html
- 大肠杆菌血清分型、志贺毒素分型和病理分型(ECTyper):https://cloud.mimazi.net/tool/article-196.html
三、SerotypeFinder小工具的使用方法
输入文件:
全基因组数据或包含抗原基因的FASTA格式文件
注意文件名禁止包含空格(如:EC Strain1.fasta,可将其修改为 EC_Strain1.fasta)
输出结果:
--output.tsv:血清型预测结果

- gene:匹配的血清型特异性基因名称;
- serotype:预测的血清型类型;
- identity:序列比对相似度(%)。

软件版本:SerotypeFinder2.0.1
数据库版本:1.0.0 (2022-05-16)
四、ECTyper小工具的使用方法
输入数据:
基因组预组装结果(FASTA格式),支持提交单个基因组或包含多个目标基因组的文件夹
注意文件名禁止包含空格(如:EC Strain1.fasta,可将其修改为 EC_Strain1.fasta)
输出结果:
--blastn_output_alleles.txt:O和 H抗原数据库的 BLASTN比对结果
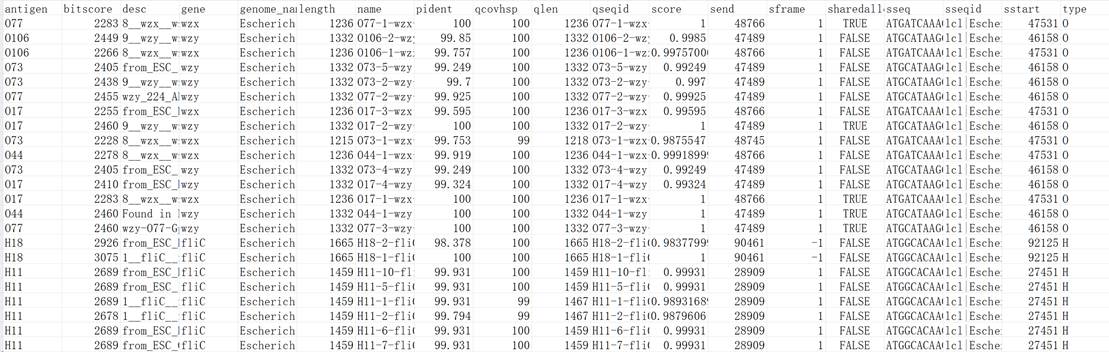
--output.tsv:血清型预测结果
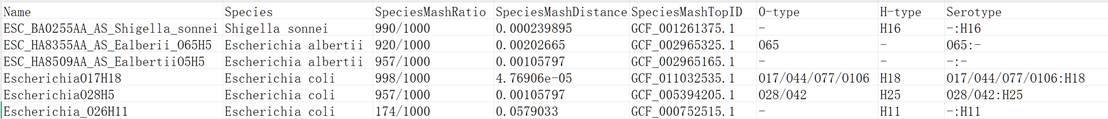
- Name:样本名称(通常为唯一标识符);
- Species:物种信息(用于识别样本污染或标签错误);
- O-type:O抗原分型;
- H-type:H抗原分型;
- Serotype:血清型预测结果(包含O和H抗原)。
软件版本:Ectyper 2.0.0
数据库版本:database version 1.0
五、参考文献:
[1] Camacho C, Coulouris G, Avagyan V, et al. BLAST+: architecture and applications. BMC Bioinformatics. 2009;10:421.
[2] Clausen PTLC, Aarestrup FM, Lund O. Rapid and precise alignment of raw reads against redundant databases with KMA. BMC Bioinformatics. 2018;19(1):307.
[3] Joensen KG, Tetzschner AM, Iguchi A, Aarestrup FM, Scheutz F. Rapid and Easy In Silico Serotyping of Escherichia coli Isolates by Use of Whole-Genome Sequencing Data. J Clin Microbiol. 2015;53(8):2410-2426.
[4]Bessonov K, Laing C, Robertson J, et al. ECTyper: in silico Escherichia coli serotype and species prediction from raw and assembled whole-genome sequence data. Microb Genom. 2021;7(12):000728.
-
点赞 (0人)
- 收藏 (0人)
-
上一篇: 没有了
- 下一篇: Easyfig罕见问题1



