实用组学小工具——轻松搞定BLAST比对
- 看不见的线
- 1572
- 2025-08-28 10:16:38
- 原创
一、关于BLAST比对
BLAST全称为Basic Local Alignment Search Tool,即基于局部序列比对算法的搜索工具,是由美国国家生物技术信息中心(National Center for Biotechnology Information, NCBI)开发和管理的一套生物大分子一级结构序列比对程序。程序可将输入的核酸碱基或蛋白质氨基酸序列与数据库中的已知来源序列进行比对,输出序列之间的同源性信息,从而辅助判断输入的序列来源或与已知序列的进化关系。
BLAST提供在线版和本地版两种使用方式:
1、在线BLAST分析:适用于查询少量序列;
2、本地BLAST分析:适用于查询还未提交至数据库的序列以及大量序列;
因此,当我们要查询大量序列时推荐使用本地版BLAST。(推荐学习课程:《个人电脑上如何通过BLAST软件做比较基因组学分析》https://college.mimazi.net/course/article-32.html)
二、零基础做生信
除此之外,还可以使用密码子·生信云平台提供在线的BLAST比对分析(自建数据库)小工具(https://cloud.mimazi.net/tool/article-22.html),用户上传fasta格式的待查询序列文件和参考序列文件,即可完成序列比对,并获得结果表。
本工具支持以下功能模块:
"blastn:核酸-核酸":BLASTN是核酸序列到核酸库中的一种查询。库中存在的每条已知序列都将同所查序列作一对一地核酸序列比对。
"blastx:核酸-蛋白":BLASTX是核酸序列到蛋白库中的一种查询。先将核酸序列翻译成蛋白序列(一条核酸序列会被翻译成可能的六条蛋白),再对每一条作一对一的蛋白序列比对。
"tblastn:蛋白-核酸":TBLASTN是蛋白序列到核酸库中的一种查询。与BLASTX相反,它是将库中的核酸序列翻译成蛋白序列,再同所查序列作蛋白与蛋白的比对。
"blastp:蛋白-蛋白":BLASTP是蛋白序列到蛋白库中的一种查询。库中存在的每条已知序列将逐一地同每条所查序列作一对一的序列比对。
密码子·生信云平台优势
零门槛体验:无需本地安装Linux环境,无需复杂配置,打开网页即可一键上传数据,轻松运行各种分析。
高效安全:云端计算资源强大,数据处理速度快,保障数据安全与隐私。
友好界面:操作界面简洁直观,适合各类用户,无需编程基础。
专业支持:提供详细的操作指南和技术支持,助力您的科研工作顺利进行。
小工具链接:https://cloud.mimazi.net/tool/article-22.html
小工具用法:
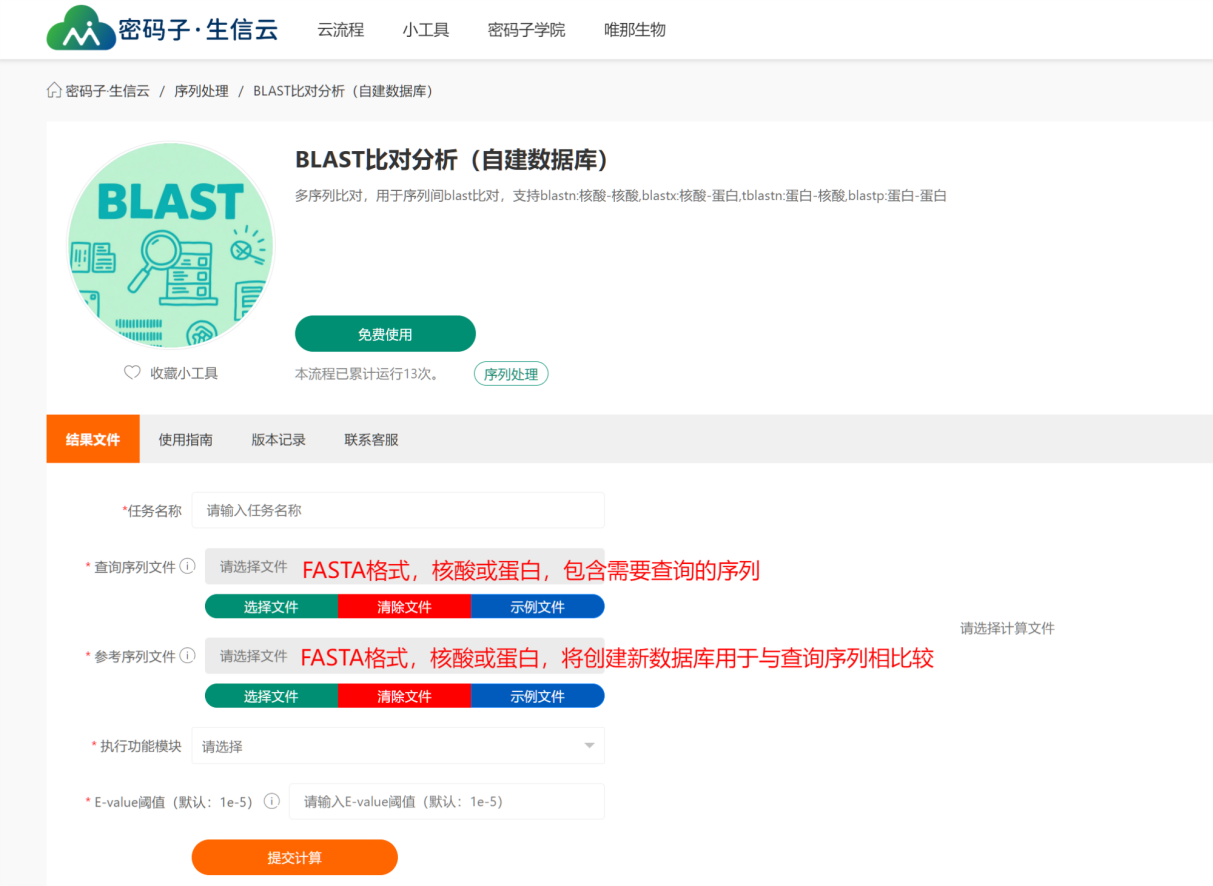
查询序列文件和参考序列文件名禁止包含空格,若文件名中包含空格,可将空格修改为“_”,(如:EC Strain1.fasta ,可将其修改为 EC_Strain1.fasta)
小工具结果:
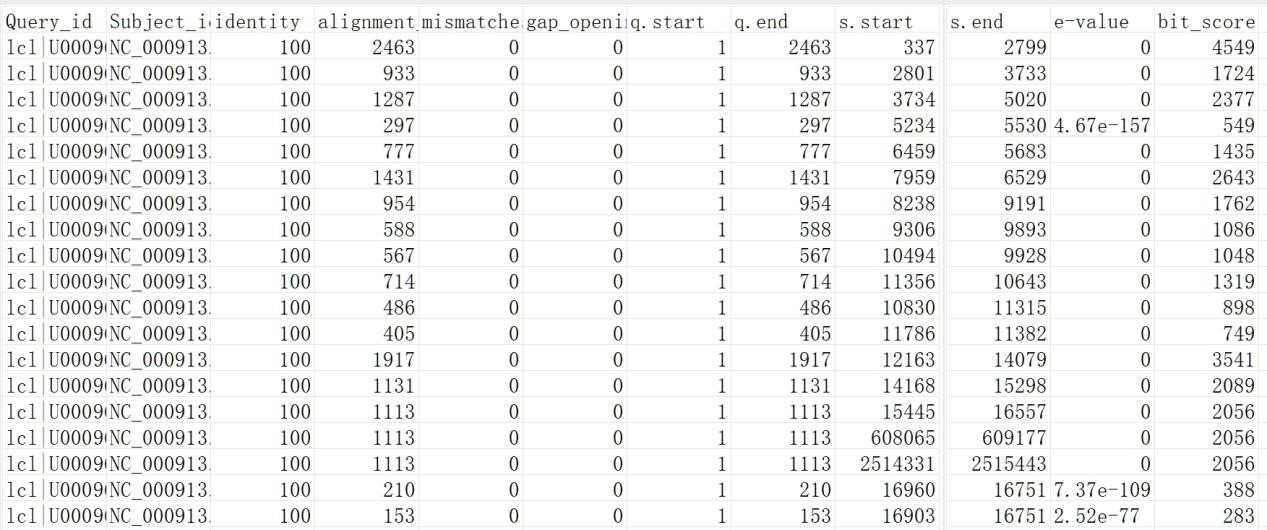
总共对应12列结果,每一列的含义如下:
query id:查询序列ID标识;
refer id:参考序列ID标识;
identity (%):序列比对的一致性百分比;
alignment length:符合比对的比对区域的长度;
mismatches:比对区域的错配数;
gap openings:比对区域的gap数目;
q.start:比对区域在查询序列上(query id)的起始位点;
q.end:比对区域在查询序列上(query id)的终止位点;
s.start:比对区域在参考序列上(refer id)的起始位点;
s.end:比对区域在参考序列上(refer id)的终止位点;
e-value:比对结果的期望值,将比对序列随机打乱重新组合,与数据库进行比对,如果功能越保守,则该值越低;
bit score:比对结果的bit score值。
软件版本:BLAST 2.15.0+
密码子生信云还提供云流程和其它各色小工具分析服务:

-
点赞 (0人)
- 收藏 (0人)



