Easyfig-genbank文件注释信息的修改或添加
- 看不见的线
- 1735
- 2023-11-23 14:05:17
- 原创
很多时候,由于基因预测或注释的问题,我们不得不修改或添加genbank文件的部分注释信息(Annotation)。总的来说,主要可以分为以下的三种情况:基因/CDS注释有误、基因/CDS预测有误、基因/CDS预测结果有缺漏。前两种情况按正常的方式直接修改即可,但第三种情况由于需要额外添加内容,我们就要注意不能影响genbank文件本身的格式,否则可能对genbank的使用造成影响。
第一种情况:基因/CDS的注释有误
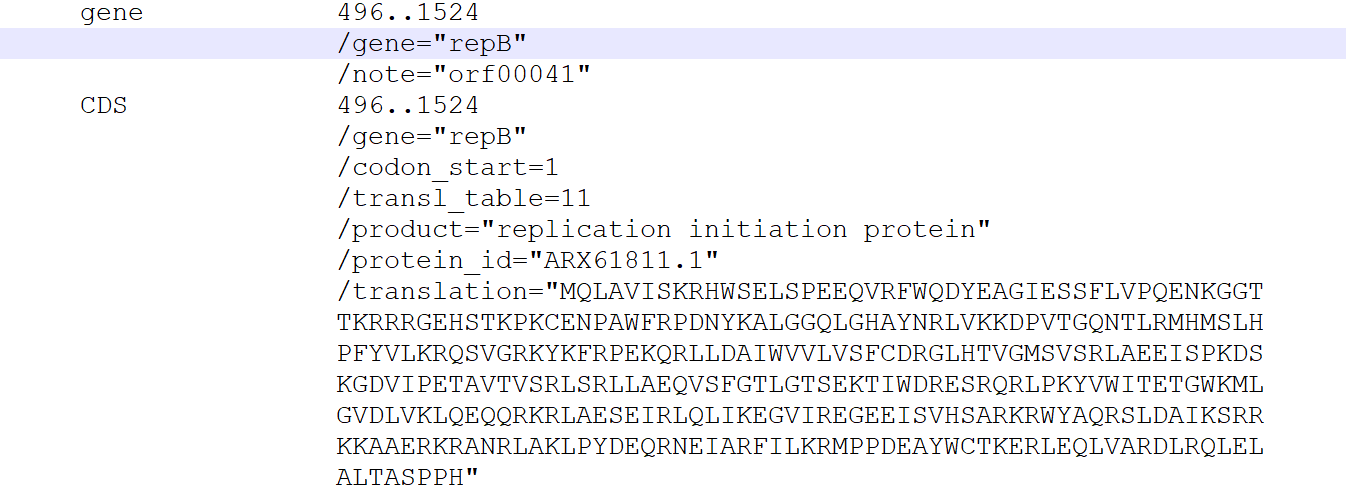
如图1和2,假设图中的基因不是repA而是repB,我们可以直接将“repA”修改为“repB”,其它内容不做变更。

第二种情况:基因/CDS预测位置有误
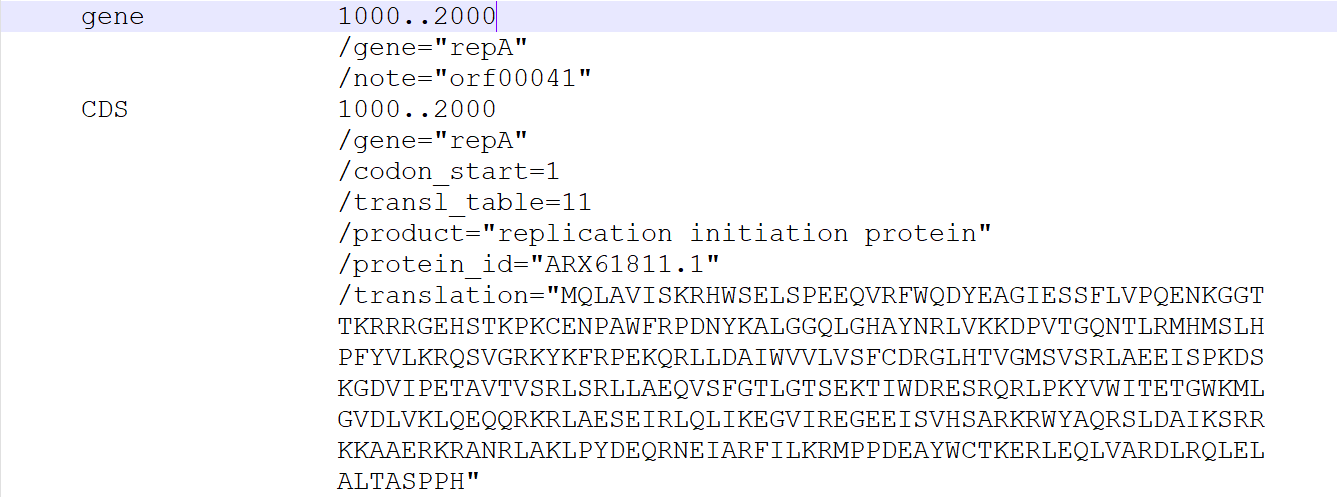
如图3和4,假设repA基因在序列中的实际位置不是496~1524 bp,而是1000~2000 bp,我们可以直接在genbank文件中将“496..1524”修改为“1000..2000”,同时对蛋白序列(translation)进行相应的替换(对于easyfig而言是否替换蛋白序列无所谓)。

第三种情况:基因/CDS预测结果有缺漏
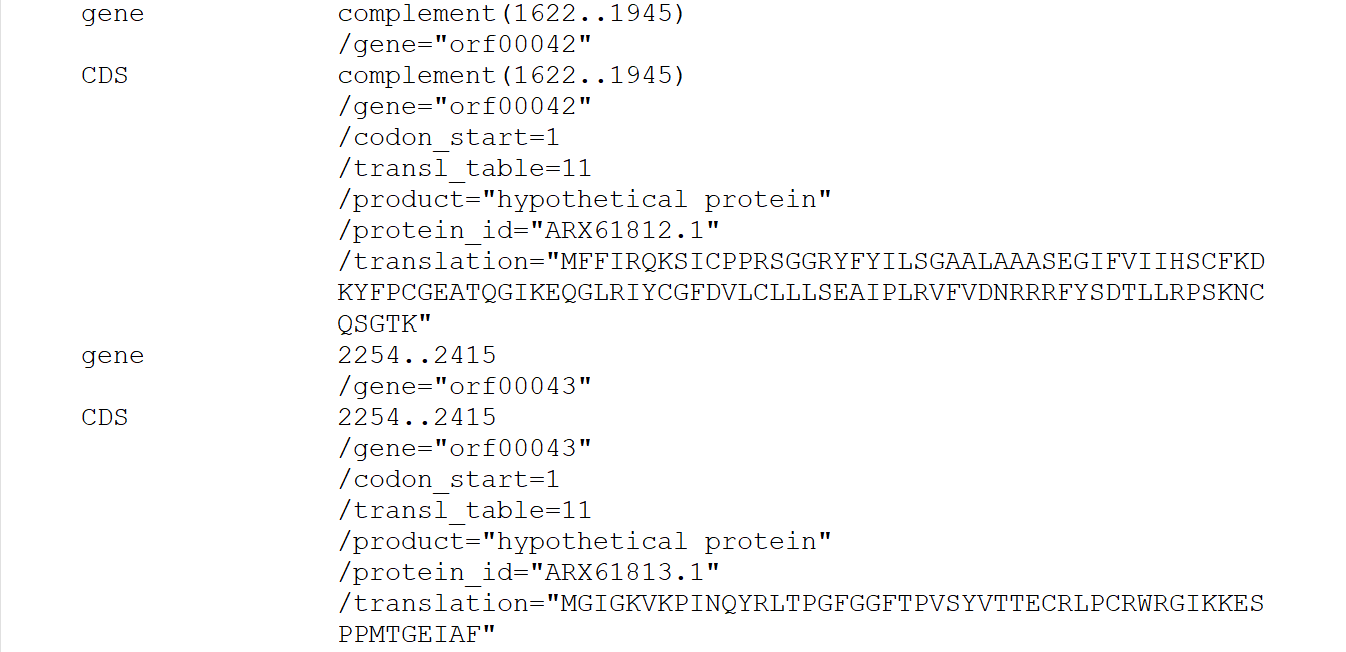
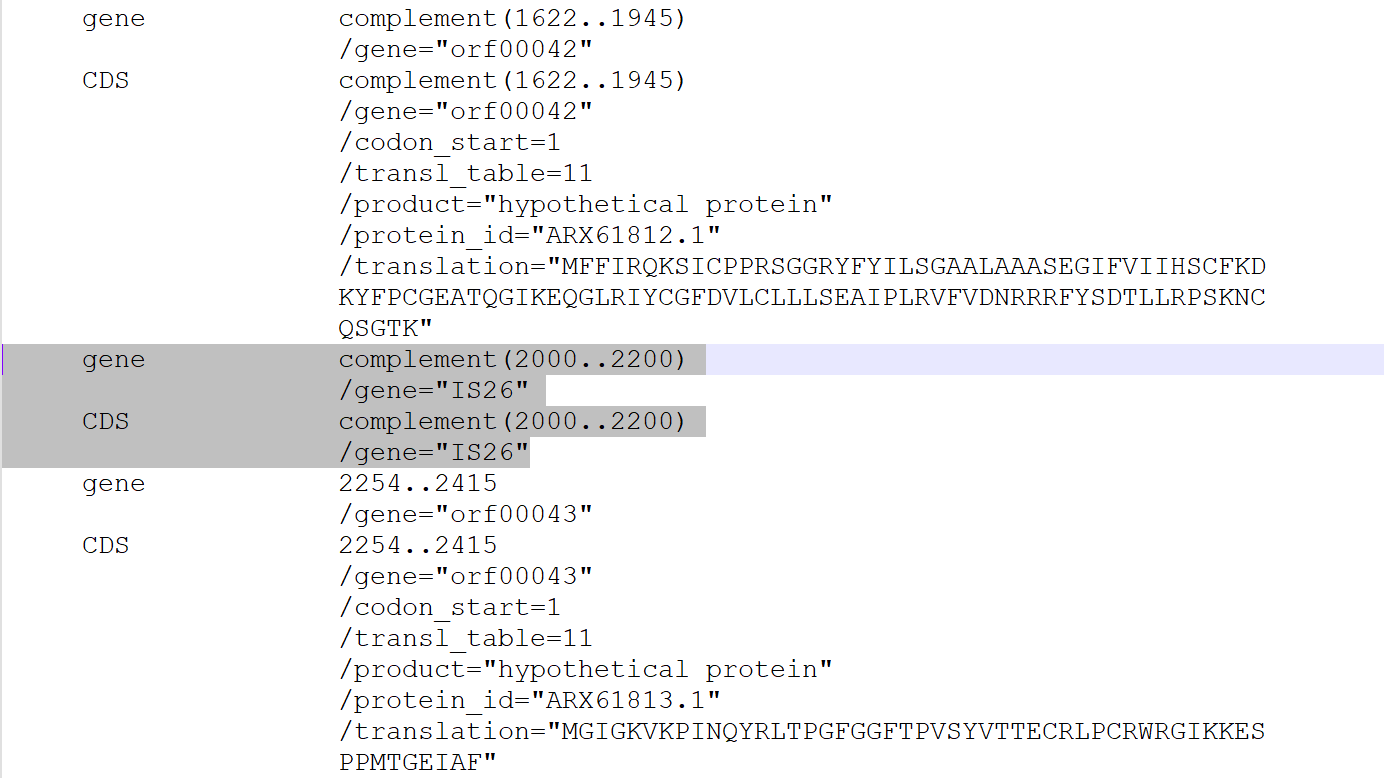
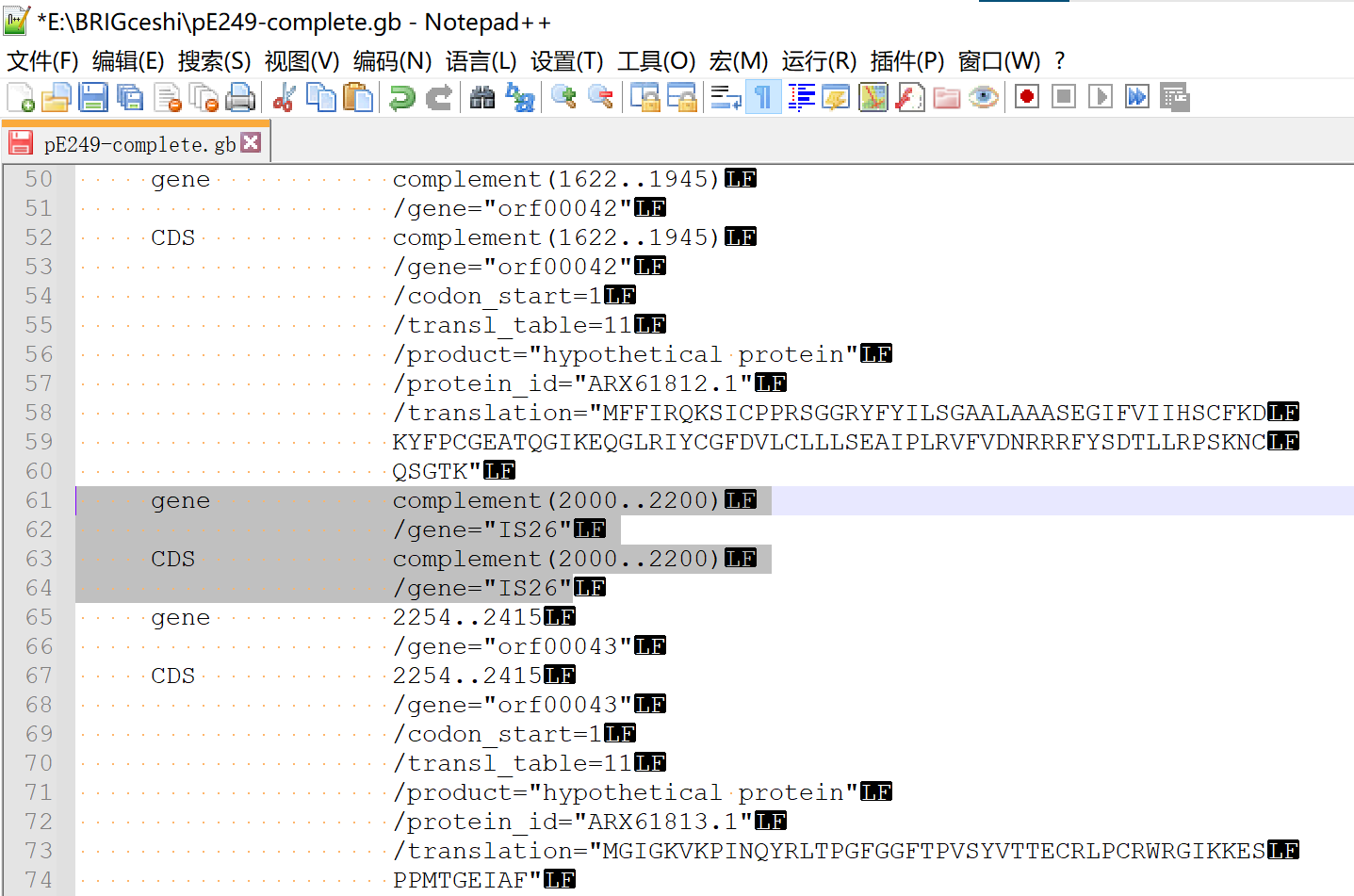
如图5,假设orf00042和orf00043之间还存在一个基因未被注释出来(2000~2200 bp)。我们可以自己添加这个基因的注释结果,常规的方法就是拷贝一个其它基因的注释信息,并修改相应的内容,包括位置信息、gene名称、蛋白序列等等(对于easyfig而言只有位置信息和gene名称是必须的,其它无所谓)。不过需要注意2点,第一点是序列有方向,所以应该确定是否需要添加“complement”;第二点是拷贝的其它基因的注释信息必须完整,所谓完整便是如图5阴影部分的状态,不能漏掉左边gene和CDS部分的任何一个空格,否则便会使得genbank文件的注释部分出错(在easyfig中这种错误可能会导致该基因及后续的基因无法正常显示)。
这么做的原因主要是因为我们用Enter新增一行时,左侧gene/CDS附近的空白处默认为Tab符,而不是genbank正常应该用到的空格符。如图6所示我在orf00042和orf00043之间添加了一个基因的注释结果,假设这个基因为IS26。正确和错误的添加方式在Notepad++软件下直接看到的都是类似的情况,但如果我们在Notepad++上方菜单栏选择“显示所有字符”,就会发现看到正常情况下左侧gene/CDS附近为一个个点,代表空格(图7),而错误添加如图8所示,在新增行的相应位置直接输入文本,可以看到左侧gene/CDS附近为箭头符号,代表Tab符。这种错误通过记事本打开genbank文件,找到添加的位置,也可以明显地看到问题。
此外,我们不增加新的gene/CDS,而是在原有基因注释信息的基础上添加一项新的内容,也会遇到这样的情况,比如我们要对orf00042基因进行颜色的注释,就可以在translation注释末尾回车后拷贝一整行类似的注释信息,比如/gene=“orf00042”及其前方的所有空格,然后进行修改,若是我们直接输入,也是犯了同样的错误(参见图9和图10)。

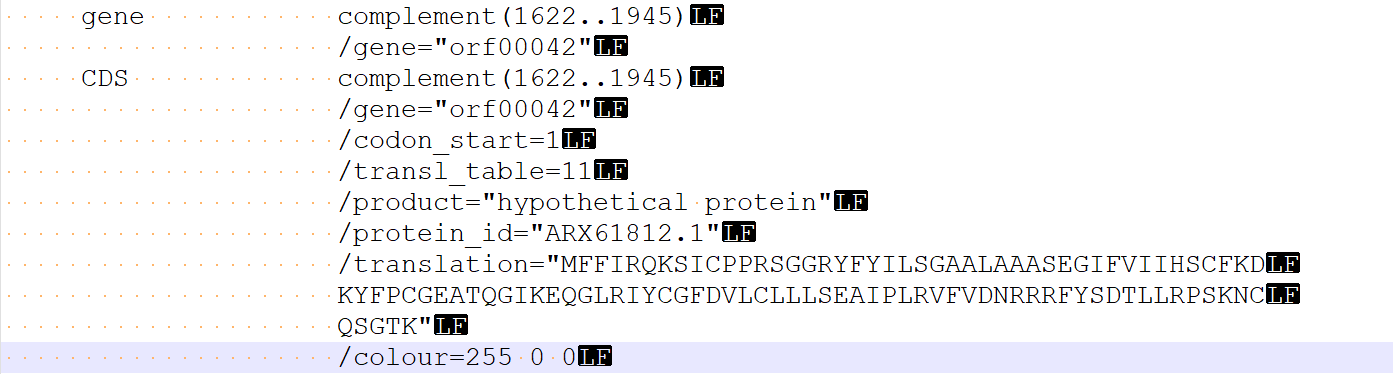

此外,注释信息的修改不影响genbank的序列部分(Origin),在easyfig中,研究特定的区域可以基于easyfig本身的功能,也可以在genbank文件中直接删掉研究区域以外的注释信息。但无论我们如何更改,都不需要,也不可以更改genbank的序列部分,否则会对最终的结果产生影响。
推荐课程
【课程】微生物比较基因组精品系列课——全套自学必入的系统课程
课程链接:微生物比较基因组精品系列课【全套】
【课程】微生物比较基因组与群体进化——基因组变异专题研究
课程链接:微生物比较基因组与群体进化
【课程】微生物分子分型-MLST课程——分型全套(含理论、软件、方法)
课程链接:微生物分子分型-MLST课程
【课程】基因组结构分析神器Easyfig实操精品课
【课程】BRIG绘图——结构比较专题2
【课程】肺炎克雷伯菌基因组学研究综合指南
课程链接:肺炎克雷伯菌基因组学研究综合指南
【课程】微生物基因组生信必学课程
课程链接:微生物基因组生信分析必学课程
【课程】微生物生防菌研究
课程链接:生防菌的系统化研究
专题材料
【资料】耐药专题材料
【资料】生防专题材料
请添加唯那生物技术客服小唯的微信号winnerbio01,备注“耐药专题”或“生防专题”,立马获取。
更多专题推荐
CORPORATE CULTURE
1、耐药毒力专题
-
点赞 (0人)
- 收藏 (0人)



