GrapeTree的安装及使用教程-最小生成树绘制
- 看不见的线
- 3966
- 2024-01-24 10:29:27
- 原创
GrapeTree是EnteroBase中的一个完全交互式的树可视化程序,它支持对树布局和元数据的简单操作。它使用邻接法(NJ),类似于PhyloViz的经典最小生成树算法(MSTree)或我们称之为MSTree V2的改进的最小生成树方法生成葡萄树。
- 1、数据准备
使用GrapeTree绘制最小生成树需要2份数据-Profile和 Isolate,两者都需要保存为.txt格式。其中Profile代表ST型的等位基因谱文件;Isolate代表样本信息文件。
以下为Profile和Isolate文件的基础样式:
① Profile文件需要包含菌株的名称#Name及等位基因编号。
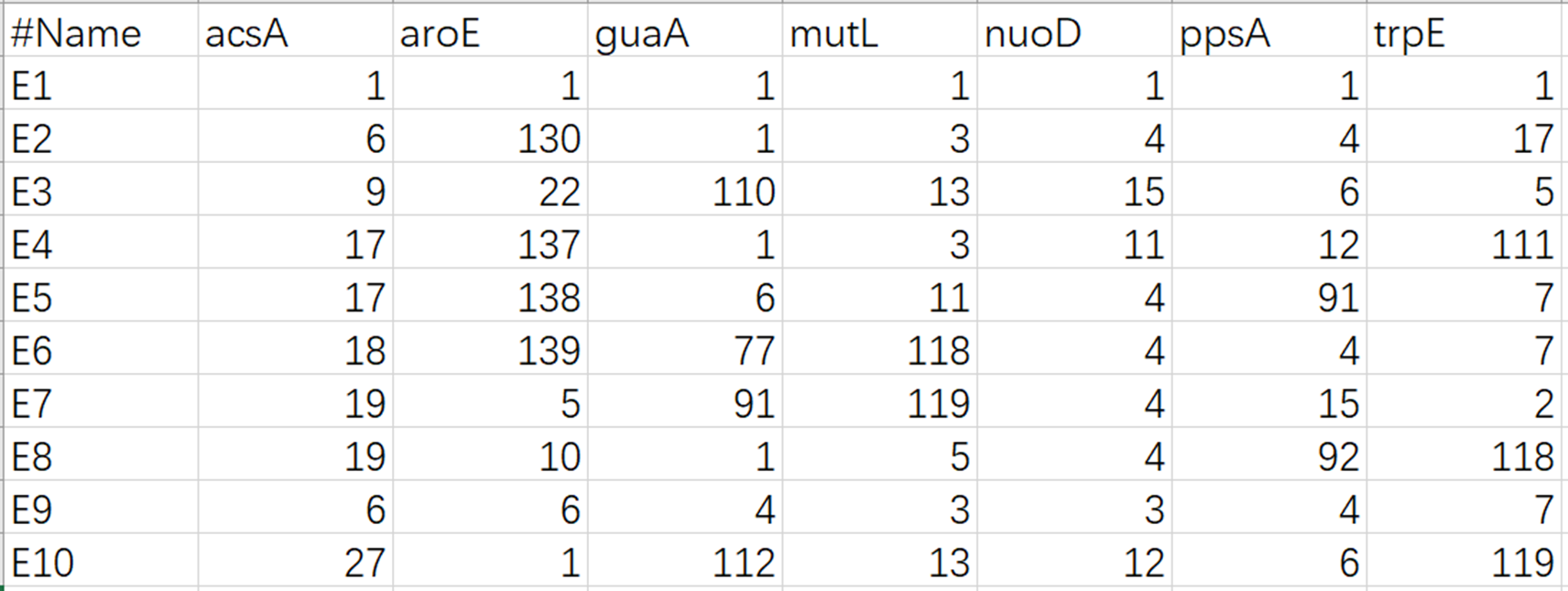
② Isolate文件可以添加分类统计的注释信息,比如分型结果ST、菌株的分离时间、分离来源、分离国家等等。

2、GrapeTree安装包下载
下载地址(https://github.com/achtman-lab/GrapeTree/releases)。
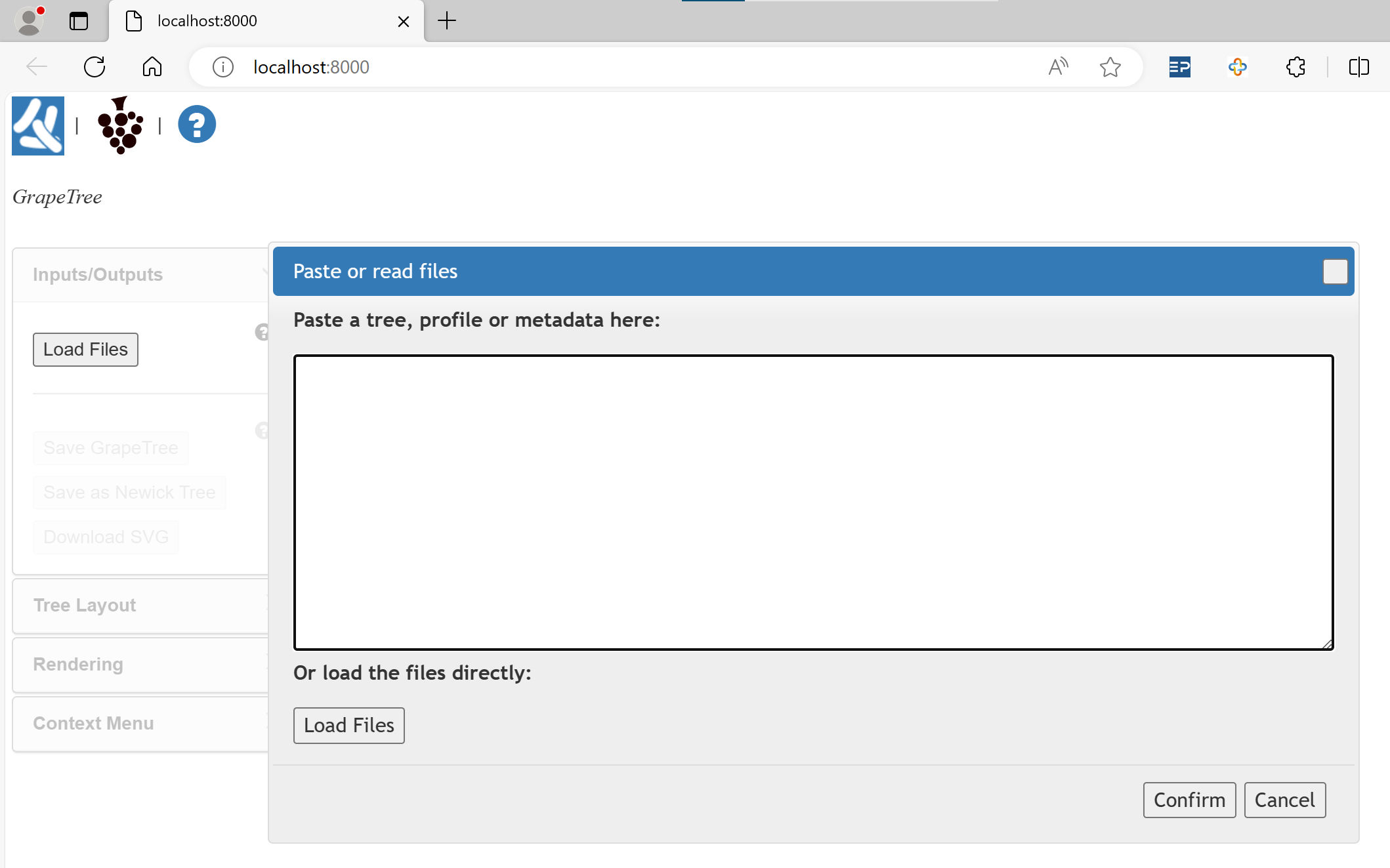
3、GrapeTree的使用说明
将下载好的GrapeTree_win.zip或GrapeTree_mac.zip进行解压,找到对应的.exe启动程序并打开。

打开GrapeTree软件后,点击Load Files,先上传Profile文件。
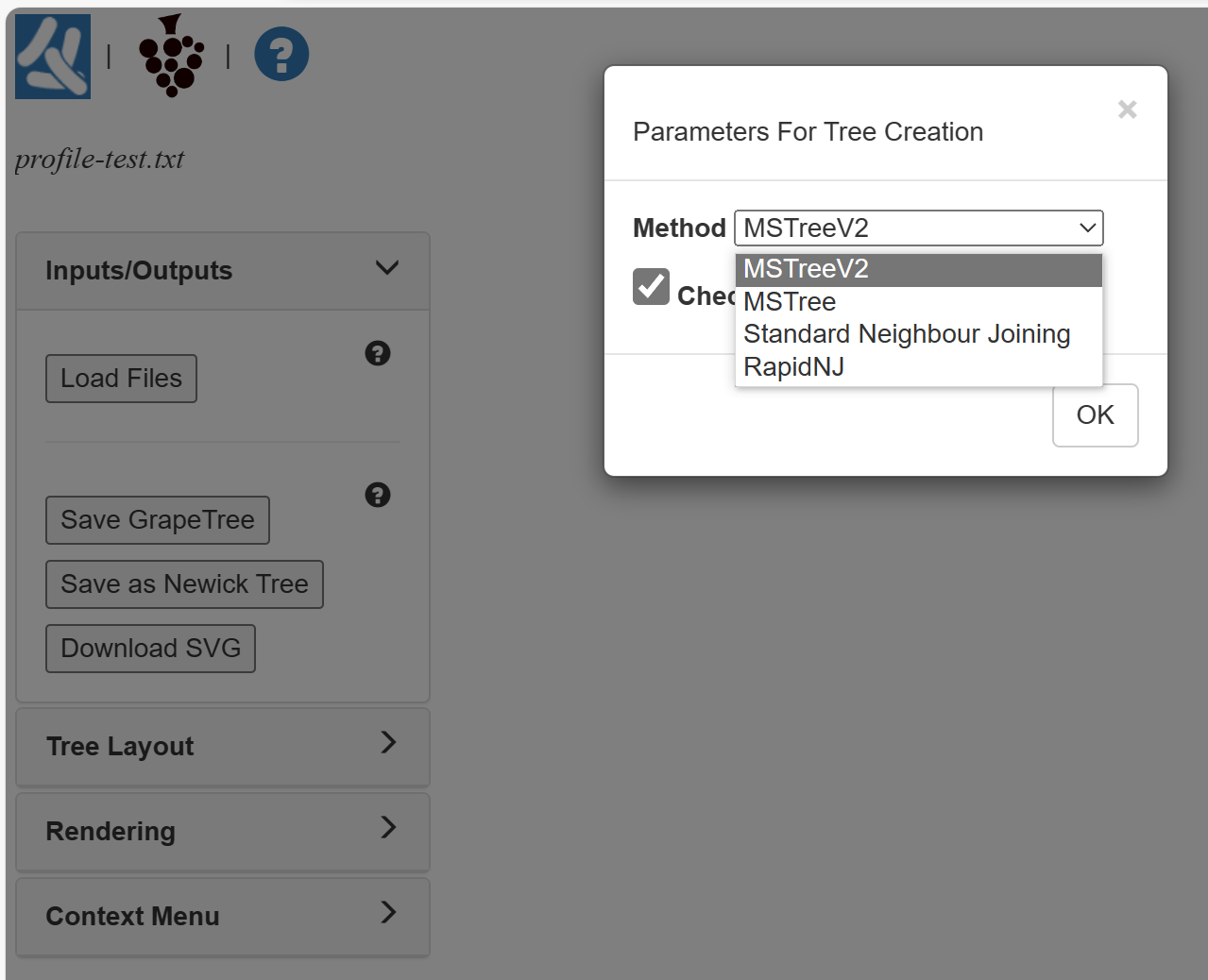
GrapeTree有以下四种建树策略可进行选择,MSTreeV2较为常用。
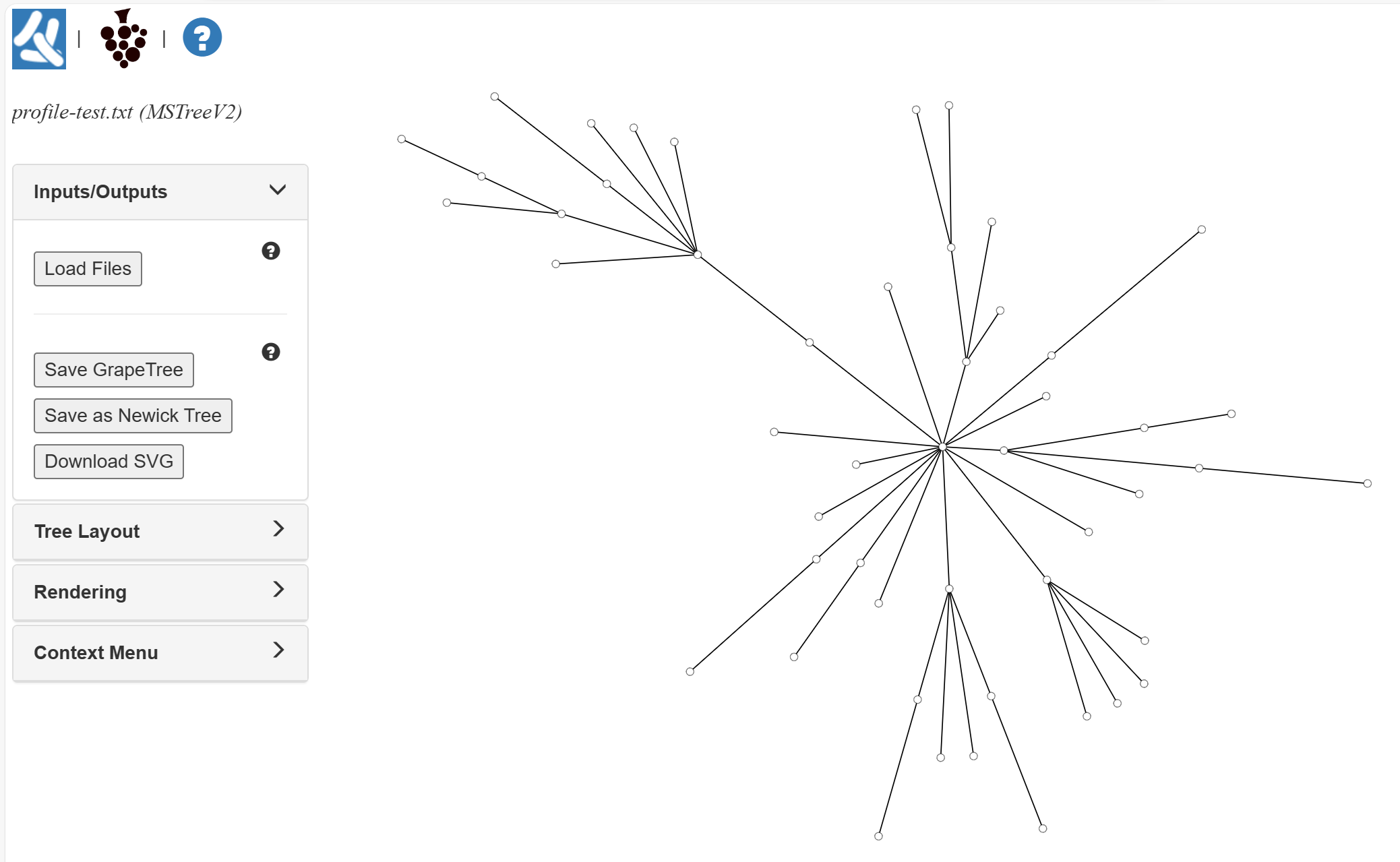
选择好建树策略后,即可生成如下图所示的基础最小生成树样式。
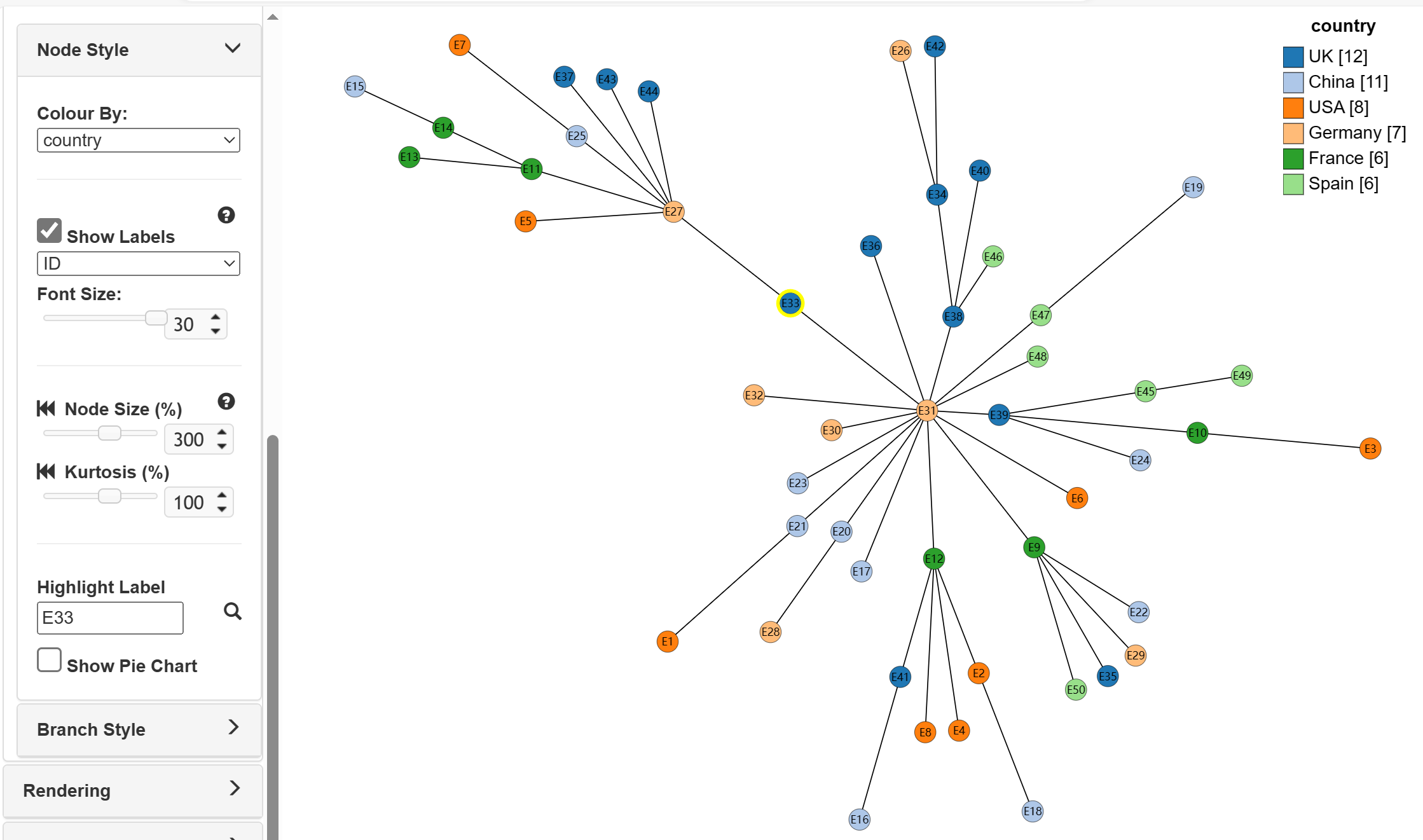
再次点击Load Files上传样本信息文件Isolate,即可添加注释信息,软件默认设置第一项注释信息,这里为“ST”。

在Tree Layout中选择Node Style-Colour By可以更换需要展示的注释信息,如将“ST”更改为“country”。

Show Labels可以选择添加样本的名称或样本对应注释信息的名称,Font Size可调节样本名称的大小,Node Size调节样本图示的大小,Highlight Label突出显示指定的样本,右侧注释标签也可以进行此操作,如此处的country处突出显示特定国家的样本。
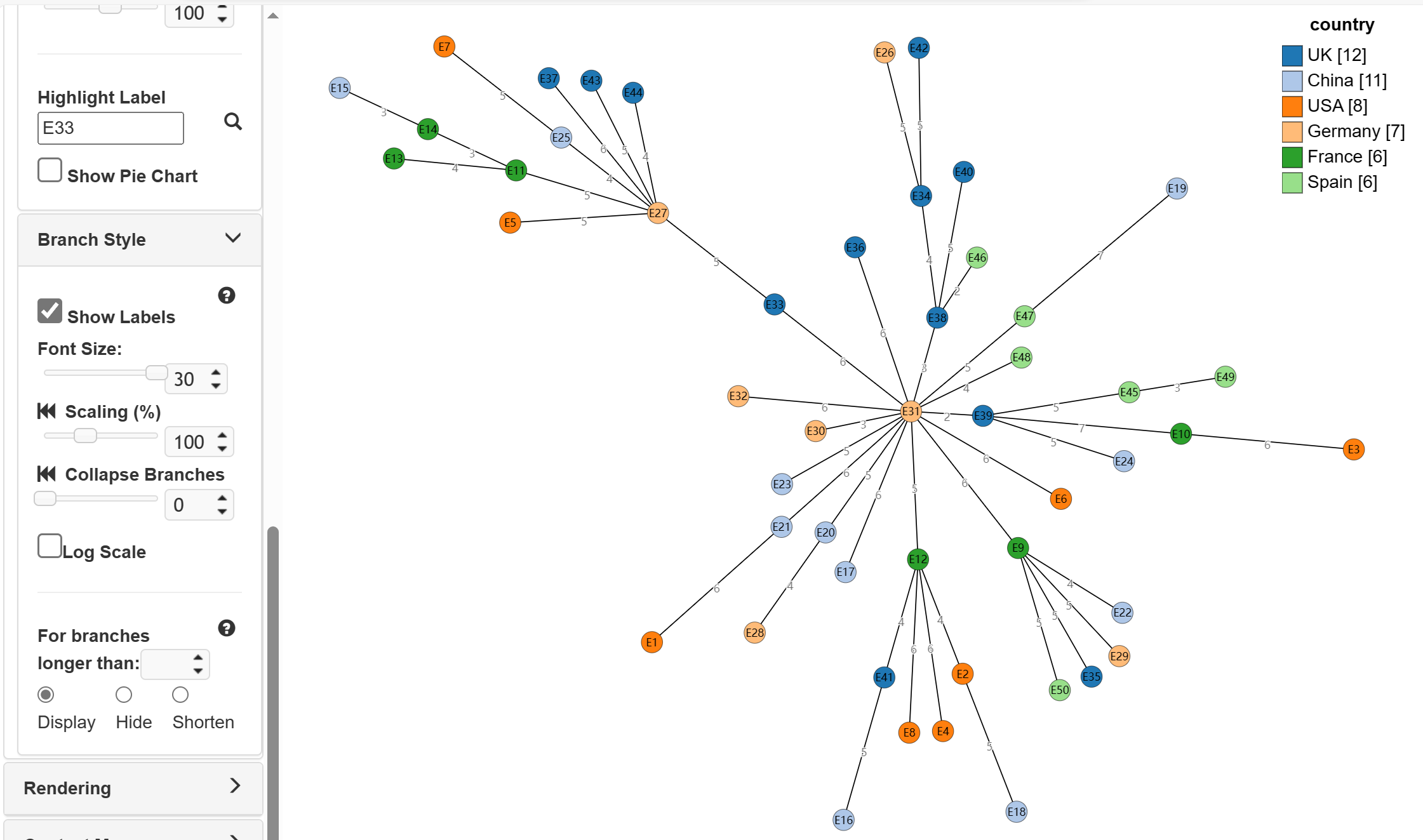
Branch Styles可以呈现等位基因的距离。
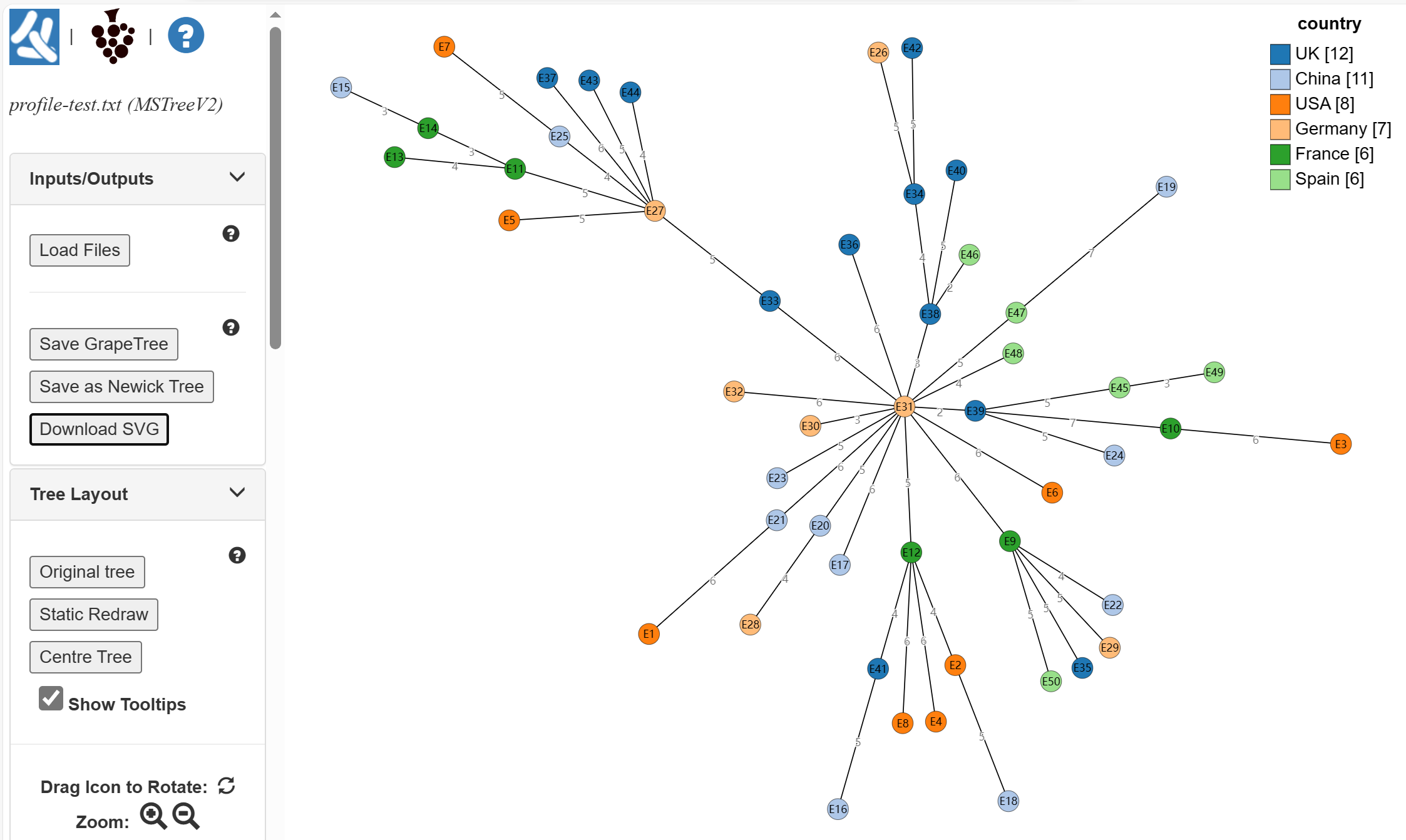
Inputs/Outputs处可保存文件或导出图像。
参考资料:
GrapeTree软件说明文档参见:
(https://enterobase.readthedocs.io/en/latest/grapetree/grapetree-tutorial-1-sa.html)
推荐课程
【课程】微生物比较基因组精品系列课——全套自学必入的系统课程
课程链接:微生物比较基因组精品系列课【全套】
【课程】铜绿假单胞菌基因组研究和分子分型实战
课程链接:铜绿假单胞菌基因组研究和分子分型实战
【课程】微生物比较基因组与群体进化——基因组变异专题研究
课程链接:微生物比较基因组与群体进化
【课程】微生物分子分型-MLST课程——分型全套(含理论、软件、方法)
课程链接:微生物分子分型-MLST课程
【课程】基因组结构分析神器Easyfig实操精品课
【课程】BRIG绘图——结构比较专题2
【课程】肺炎克雷伯菌基因组学研究综合指南
课程链接:肺炎克雷伯菌基因组学研究综合指南
【课程】微生物基因组生信必学课程
课程链接:微生物基因组生信分析必学课程
【课程】微生物生防菌研究
课程链接:生防菌的系统化研究
专题材料
【资料】耐药专题材料
【资料】生防专题材料
请添加唯那生物技术客服小唯的微信号winnerbio01,备注“耐药专题”或“生防专题”,立马获取。
更多专题推荐
CORPORATE CULTURE
1、耐药毒力专题
-
点赞 (0人)
- 收藏 (0人)



