统计基因、基因组序列长度、GC含量和GC-skew
- 看不见的线
- 560
- 2025-06-30 08:34:33
- 原创
Seqkit2是 Seqkit的新版本,是一个专门用于处理和分析生物序列数据的软件工具。它支持多种序列数据格式,包括FASTA、FASTQ等,并提供了一系列有用的功能,如数据处理、过滤、统计、格式转换等,是生物信息学领域中常用的工具之一。以下是使用seqkit统计基因、基因组序列长度、GC含量和GC-skew。
fx2tab子命令:将FASTA/Q转换为表格格式,可用于统计序列的信息
# Usage: seqkit fx2tab [flags]
# flags:
-a, --alphabet:打印字母表字母。
-q, --avg-qual:打印读取的平均质量。
-B, --base-content strings:打印碱基含量。支持多个值,例如-B AT -B N。
-C, --base-count strings:打印碱基计数。支持多个值,例如-C AT -C N。
-I, --case-sensitive:计算区分大小写的碱基含量/序列哈希。
-g, --gc:打印 GC含量。
-G, --gc-skew:打印 GC-Skew。
-H, --header-line:打印标题行。
-l, --length:打印序列长度。
-n, --name:仅打印名称(不包括序列和质量)。
-Q, --no-qual:即使对于 FASTQ文件,也仅输出两列。
-i, --only-id:打印ID而不是完整标题。
-b, --qual-ascii-base int: ASCII基数,Phred+33为33(默认为33)。
-s, --seq-hash:打印序列的哈希(MD5)。
#帮助信息查看
seqkit fx2tab -h
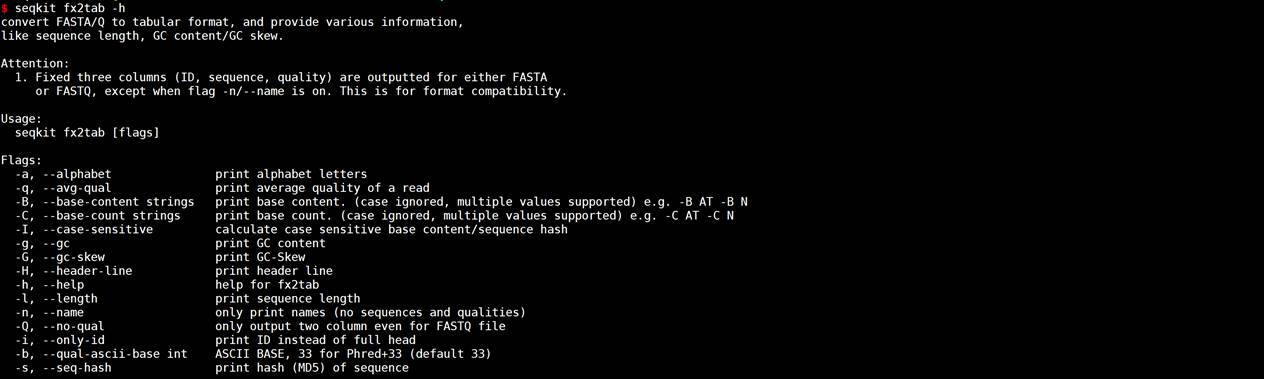
#打印序列长度、GC含量,并输出前10行
seqkit fx2tab viral.1.1.genomic.fna.gz -l -g -n -i -H | head

#打印序列长度、GC含量,将统计结果导出为制表符分隔文件
seqkit fx2tab viral.1.1.genomic.fna.gz -l -g -n -i -H > viral.1.1.genomic.fna_length_GC.tsv

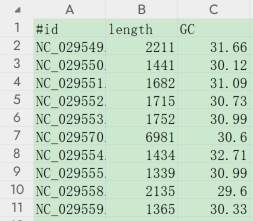
# -l统计序列长度
# -g统计平均GC含量
# -i只打印名称(不打印序列)
# -H打印标题行
-
点赞 (0人)
- 收藏 (0人)



