Canu安装及使用教程 ——基因组三代数据组装软件
- 看不见的线
- 3505
- 2024-01-11 08:47:53
- 原创
Canu是一款使用广泛的三代组装软件,可用于组装PacBio或Oxford Nanopore序列,也支持pacbio hifi的组装。Canu对PacBio和Oxford Nanopore原始数据的组装分为三个步骤:纠错,修整和组装。
每一步经历以下几个步骤:
1.加载read到read数据库(seqStore);
2.进行k-mer计数;
3.计算overlap,加载到数据库(OvlStore);
4.根据overlap进行纠错/修剪/组装。

1、Canu的安装
二进制发行版本安装
https://github.com/marbl/canu/releases
#下载可执行文件并提取文件
curl -L https://github.com/marbl/canu/releases/download/v2.2/canu-2.2..Linux.tar.xz --output canu-2.2..Linux.tar.xz
tar -xJf canu-2.2.*.tar.xz
# 切换到bin目录
cd canu-2.2/bin/
# 添加环境变量(临时)
export PATH=$PATH:$(pwd)
2、Canu的使用
#查看帮助信息
canu -h
#运行(PacBio测序数据)
canu -p ecoli -d test genomeSize=4.8m -pacbio pacbio.fastq
#运行(nanopore测序数据)
canu -p ecoli -d test genomeSize=4.8m maxInputCoverage=100 -nanopore nanopore.fastq
参数说明:
-p:组装输出文件前缀
-d:组装文件输出文件夹
genomeSize=4.8m:指定待组装基因组的大小。这里设置为 4.8m,表示预估的基因组大小为 4,800,000个碱基对。
-pacbio-raw:指使用pacbioCLR原始数据
-nanopore-raw:指使用nanopore原始数据
-pacbio-hifi:指使用pacbio-hifireads进行组装
rawErrorRate:未纠错read之间允许的最大差异碱基数,默认PacBioreads为0.300,Nanoporereads为0.500
correctedErrorRate:纠错后read之间允许的最大差异碱基数,默认PacBioreads为0.045,Nanoporereads为0.144
corOutCoverage:用于纠错的数据最小coverage,默认是40x
minReadLength:使用长度大于该阈值的reads,默认为1000
minOverlapLength:最小overlap的长度,默认为500
maxThreads:设置运行的最大线程数
- 结果说明
Canu运行后生成的全部结果在test目录下,内容如下:
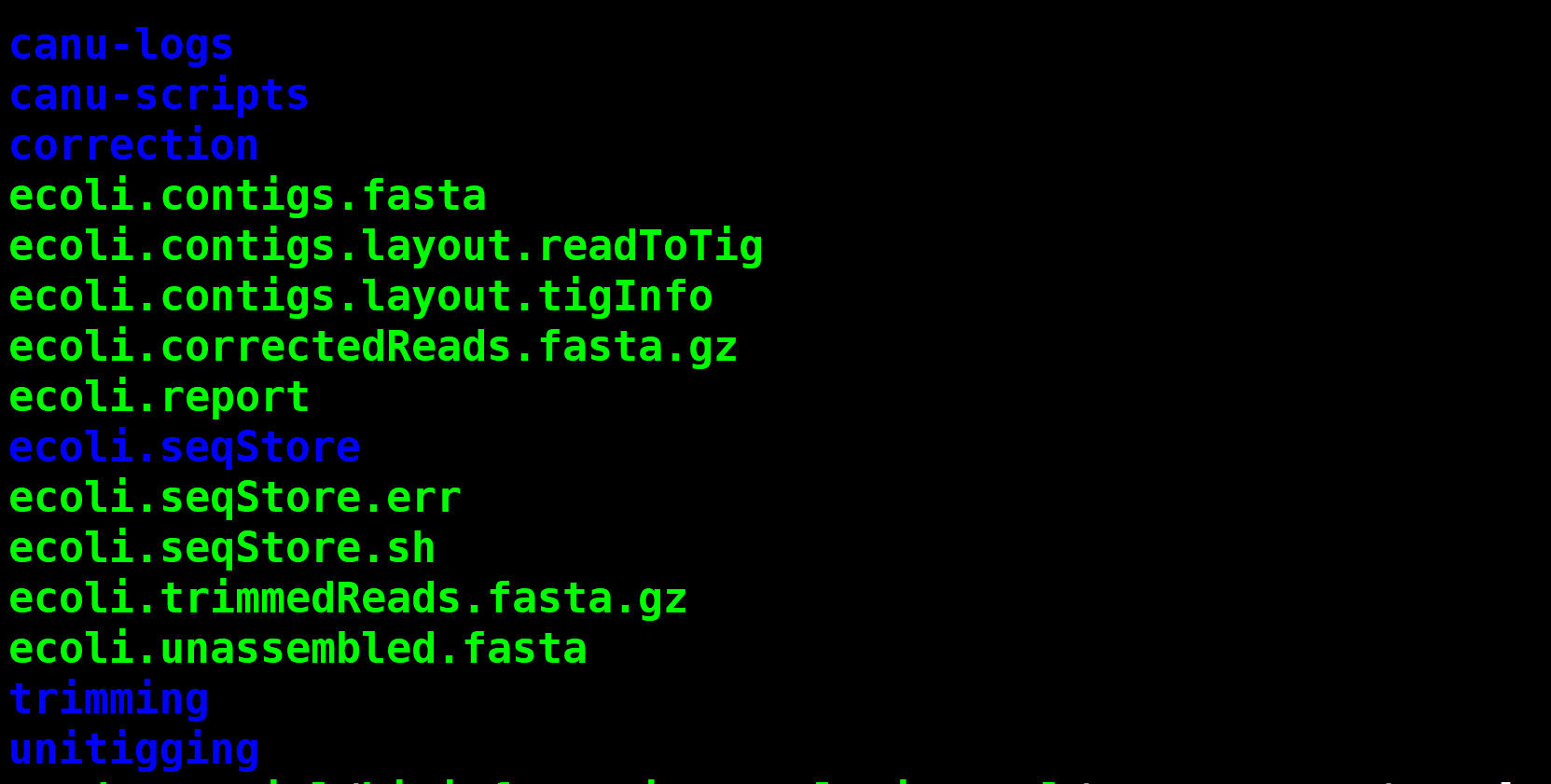
correction,trimming,unitigging记录了纠错、修剪和组装的过程,主要结果文件包括:
ecoli.correctedReads.fasta.gz:纠错后的reads
ecoli.trimmedReads.fasta.gz:修剪后的reads
ecoli.contigs.fasta:最终的组装contigs
ecoli.report:记录整个Canu运行过程及结果
参考资料:
Koren S, Walenz BP, Berlin K, Miller JR, Phillippy AM. Canu: scalable and accurate long-read assembly via adaptive k-mer weighting and repeat separation. Genome Research. (2017). doi:10.1101/gr.215087.116
Koren S, Rhie A, Walenz BP, Dilthey AT, Bickhart DM, Kingan SB, Hiendleder S, Williams JL, Smith TPL, Phillippy AM. De novo assembly of haplotype-resolved genomes with trio binning. Nature Biotechnology. (2018).
Nurk, S., Walenz, B.P., Rhie, A., Vollger, M.R., Logsdon, G.A., Grothe, R., Miga, K.H., Eichler, E.E., Phillippy, A.M., and Koren, S. (2020). HiCanu: accurate assembly of segmental duplications, satellites, and allelic variants from high-fidelity long reads. Genome Res 30, 1291-1305.
推荐课程
【课程】微生物比较基因组精品系列课——全套自学必入的系统课程
课程链接:微生物比较基因组精品系列课【全套】
【课程】铜绿假单胞菌基因组研究和分子分型实战
课程链接:铜绿假单胞菌基因组研究和分子分型实战
【课程】微生物比较基因组与群体进化——基因组变异专题研究
课程链接:微生物比较基因组与群体进化
【课程】微生物分子分型-MLST课程——分型全套(含理论、软件、方法)
课程链接:微生物分子分型-MLST课程
【课程】基因组结构分析神器Easyfig实操精品课
【课程】BRIG绘图——结构比较专题2
【课程】肺炎克雷伯菌基因组学研究综合指南
课程链接:肺炎克雷伯菌基因组学研究综合指南
【课程】微生物基因组生信必学课程
课程链接:微生物基因组生信分析必学课程
【课程】微生物生防菌研究
课程链接:生防菌的系统化研究
专题材料
【资料】耐药专题材料
【资料】生防专题材料
请添加唯那生物技术客服小唯的微信号winnerbio01,备注“耐药专题”或“生防专题”,立马获取。
更多专题推荐
CORPORATE CULTURE
1、耐药毒力专题
-
点赞 (0人)
- 收藏 (0人)



